
Unsere Forschung
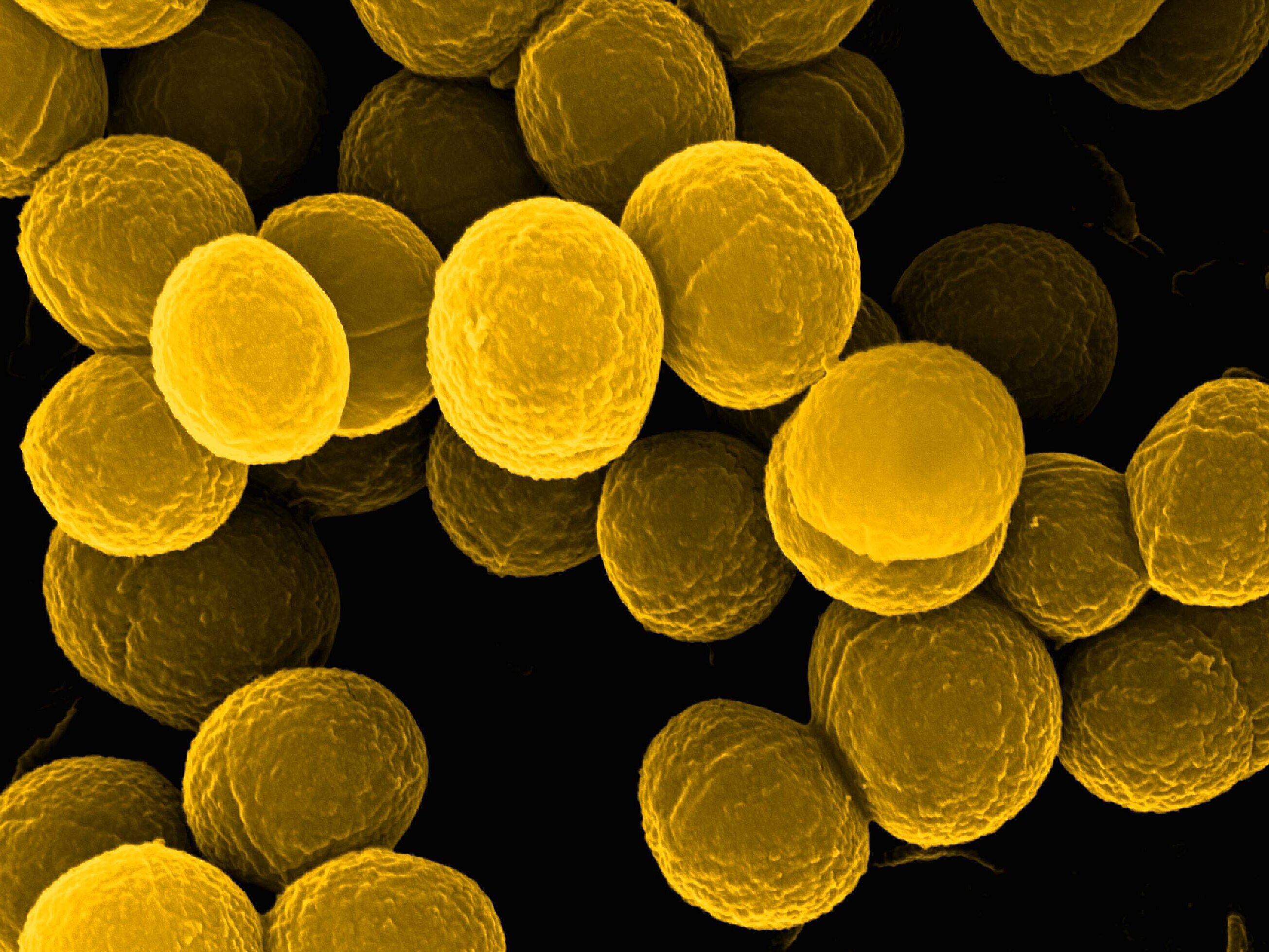
Die wichtigsten Untersuchungsobjekte unserer Forschungsgruppe sind die humanpathogenen Bakterien Staphylococcus aureus und Pseudomonas aeruginosa. Diese Bakterien gehören weltweit zu den gefürchtetsten humanpathogenen Erregern und sind für mehr als die Hälfte aller Krankenhausinfektionen verantwortlich. Besonders alarmierend ist in diesem Zusammenhang die steigende Zahl von Infektionen mit multiresistenten Erregerstämmen, die auf eine Behandlung mit herkömmlichen Antibiotika nicht mehr ansprechen und daher häufig zum Tod des Patienten führen.
Daher ist es wichtig, neue Behandlungsstrategien für diese Erreger zu entwickeln, die sich leicht in der klinischen Praxis leicht anwendbar sind. Um dies zu erreichen, ist ein umfassendes und tiefes Verständnis der Pathophysiologie und Virulenz dieser Erreger dringend erforderlich - daher konzentrieren wir uns insbesondere auf die Aufklärung des Zusammenhangs zwischen der Physiologie der Erreger und ihrer Fähigkeit, ihren Wirt zu kolonisieren und Krankheiten zu verursachen.
Die eigentlichen Effektoren in einer Zelle sind Proteine, die für die Interaktion des Bakteriums mit seinem Wirt von entscheidender Bedeutung sind. Wir wollen verstehen, welche Prozesse in Bakterien sich bei einer Infektion verändern, welche davon essentiell sind und welche Proteine daran beteiligt sind. Wir sind nicht nur daran interessiert, die Synthese und den Abbau von Proteinen in Bakterienzellen systematisch zu untersuchen, sondern wir versuchen auch, die Aktivität und Modifikation dieser Moleküle sowie die Funktionen, die sie während der Interaktion dieser Bakterien mit ihrem Wirt erfüllen, aufzuklären. Wir konzentrieren uns insbesondere auf sehr kleine Proteine mit bis zu 100 Aminosäuren, die bisher nur wenig charakterisiert sind.
Um die Menge, den Abbau und die Interaktion von Proteinen in bakteriellen und eukaryotischen Zellen unter Infektionsbedingungen sowie die Regulation und Funktion von bakteriellen Proteinen, die an der Anpassung an den Wirt beteiligt sind, systematisch zu untersuchen, haben wir verschiedene Arbeitsabläufe zur globalen Identifizierung und Quantifizierung (markierungsfrei, TMT, SILAC) von Proteinen (lösliche, sekretierte und membranassoziierte Proteine) in bakteriellen und eukaryotischen Zellen entwickelt, einen Proteogenomik-Ansatz in Kombination mit N-Terminomik zur Identifizierung nicht annotierter kleiner Proteine und Proteoformen, einen Metaproteomik-Ansatz zur Untersuchung der Physiologie bakterieller Gemeinschaften sowie Ansätze zur Identifizierung von Protein-Protein- und Protein-Metabolit-Wechselwirkungen.
Unsere Forschung
Die wichtigsten Untersuchungsobjekte unserer Forschungsgruppe sind die humanpathogenen Bakterien Staphylococcus aureus und Pseudomonas aeruginosa. Diese Bakterien gehören weltweit zu den gefürchtetsten humanpathogenen Erregern und sind für mehr als die Hälfte aller Krankenhausinfektionen verantwortlich. Besonders alarmierend ist in diesem Zusammenhang die steigende Zahl von Infektionen mit multiresistenten Erregerstämmen, die auf eine Behandlung mit herkömmlichen Antibiotika nicht mehr ansprechen und daher häufig zum Tod des Patienten führen.
Daher ist es wichtig, neue Behandlungsstrategien für diese Erreger zu entwickeln, die sich leicht in der klinischen Praxis leicht anwendbar sind. Um dies zu erreichen, ist ein umfassendes und tiefes Verständnis der Pathophysiologie und Virulenz dieser Erreger dringend erforderlich - daher konzentrieren wir uns insbesondere auf die Aufklärung des Zusammenhangs zwischen der Physiologie der Erreger und ihrer Fähigkeit, ihren Wirt zu kolonisieren und Krankheiten zu verursachen.
Die eigentlichen Effektoren in einer Zelle sind Proteine, die für die Interaktion des Bakteriums mit seinem Wirt von entscheidender Bedeutung sind. Wir wollen verstehen, welche Prozesse in Bakterien sich bei einer Infektion verändern, welche davon essentiell sind und welche Proteine daran beteiligt sind. Wir sind nicht nur daran interessiert, die Synthese und den Abbau von Proteinen in Bakterienzellen systematisch zu untersuchen, sondern wir versuchen auch, die Aktivität und Modifikation dieser Moleküle sowie die Funktionen, die sie während der Interaktion dieser Bakterien mit ihrem Wirt erfüllen, aufzuklären. Wir konzentrieren uns insbesondere auf sehr kleine Proteine mit bis zu 100 Aminosäuren, die bisher nur wenig charakterisiert sind.
Um die Menge, den Abbau und die Interaktion von Proteinen in bakteriellen und eukaryotischen Zellen unter Infektionsbedingungen sowie die Regulation und Funktion von bakteriellen Proteinen, die an der Anpassung an den Wirt beteiligt sind, systematisch zu untersuchen, haben wir verschiedene Arbeitsabläufe zur globalen Identifizierung und Quantifizierung (markierungsfrei, TMT, SILAC) von Proteinen (lösliche, sekretierte und membranassoziierte Proteine) in bakteriellen und eukaryotischen Zellen entwickelt, einen Proteogenomik-Ansatz in Kombination mit N-Terminomik zur Identifizierung nicht annotierter kleiner Proteine und Proteoformen, einen Metaproteomik-Ansatz zur Untersuchung der Physiologie bakterieller Gemeinschaften sowie Ansätze zur Identifizierung von Protein-Protein- und Protein-Metabolit-Wechselwirkungen.
Prof. Dr. Susanne Engelmann
Mit den bakteriellen Genomsequenzen wird uns der komplette Bauplan für die entsprechenden Bakterien in die Hand gegeben, dessen Komplexität und Variabilität uns immer wieder erstaunen lässt und zu neuen Fragen inspiriert.

Susanne Engelmann studierte Biologie an der Universität Greifswald und promovierte dort am Institut für Mikrobiologie. Im Jahr 2001 übernahm sie die Leitung der Arbeitsgruppe „Pathogenomik“ am Greifswalder Institut für Mikrobiologie und folgte im Jahr 2013 dem Ruf der TU Braunschweig auf die Professur für Mikrobielle Proteomforschung. Gleichzeitig übernahm sie die Leitung der Arbeitsgruppe „Mikrobielle Proteomik“ am HZI.
Team







Ausgewählte Publikationen
Froschauer, K., S. L. Svensson, R. Gelhausen, E. Fiore, P. Kible, A. Klaude, M. Kucklick, S. Fuchs, F. Eggenhofer, C. Jang, S. Engelmann, R. Backofen, C. M. Sharma. 2025. Complementary Ribo-Seq approaches map the translatome and provide a small protein census in the foodborne pathogen Campylobacter jejuni. Nat. Comm. 16:3078. doi: 10.1038/s41467-025-58329-w
Fuchs, S., M. Kucklick, E. Lehmann, A. Beckmann, M. Wilkens, B. Kolte, A. Mustafayeva, T. Ludwig, M. Diwo, J. Wissing, L. Jänsch, C.H. Ahrens, Z. Ignatova, and S. Engelmann. 2021. Towards the characterization of the hidden world of small proteins in Staphylococcus aureus, a proteogenomics approach. PloS Genetics. 17(6):e1009585. doi: 10.1371/journal.pgen.1009585.
Bartholomäus, A., B. Kolte, A. Mustafayeva, I. Goebel, S. Fuchs, D. Benndorf, S. Engelmann, Z. Ignatova. 2021. smORFer: a modular algorithm to detect small ORFs in prokaryotes. Nuc. Acid Res. 49:e89. doi: 10.1093/nar/gkab477.
Selle M., Hertlein T., Oesterreich B., Klemm T., Kloppot P., Müller E., Ehricht R., Stentzel S., Bröker B.M., Engelmann S., Ohlsen K. (2016) Global antibody response to S. aureus live-cell vaccination. Sci Rep 6:24754.
Boedeker C., Schüler M., Reintjes G., Jeske O., van Teeseling M., Jogler M., Rast P., Borchert D., Devos D.P., Kucklick M., Schaffer M., Kolter R., van Niftrik L., Engelmann S., Amann R., Rhode M., Engelhardt H., Jogler C. (2017) Determining the bacterial cell biology of Planctomycetes. Nat Commun 8:14853.


