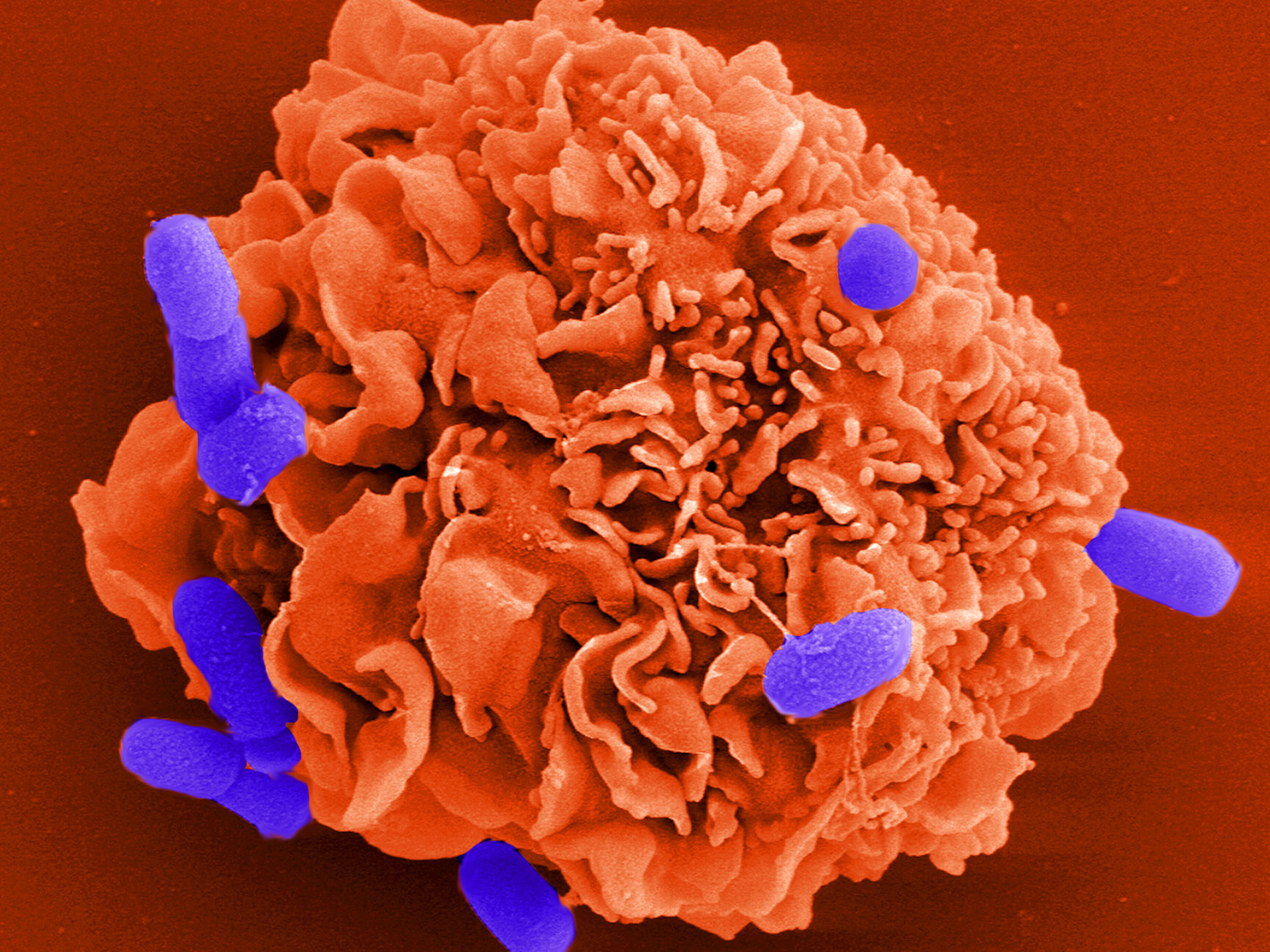

Unsere Forschung
Die aktuellen Berichte der Weltgesundheitsorganisation (WHO) mit dem Titel „The Global Burden of Disease“ (GBD) belegen deutlich die Wichtigkeit der Forschung an Wirt-Pathogen-Interaktionen. Evolution ist ein kontinuierlicher Prozess, der immer stattfindet und hochvirulente und multiresistente Bakterienstämme wie auch sogenannte „emerging pathogens“ hervorbringt. Ein tiefes Verständnis für das komplexe Wechselspiel zwischen krankheitserregenden Bakterien und dem Wirt, ist unabdingbar, um diesen Problemen in Zukunft angemessen begegnen zu können.
Kernexpertise: Kombination von Videomikroskopie und Proteinbiochemie
Die HZI-Abteilung Zellbiologie befasst sich sowohl mit den Verteidigungsmechanismen des Wirtes als auch mit den Virulenzmechanismen der Pathogene. Die Kernexpertise liegt in der Kombination aus Videomikroskopie und Proteinbiochemie, angewendet an verschiedensten Zellbewegungsmodellen und im Kontext bakterieller Infektion. In der Vergangenheit konnte die Abteilung sowohl grundlegende Regulationsmechanismen der Zellbewegung aufklären, als auch Beispiele für deren Beeinflussung durch bakterielle Virulenzfaktoren aufzeigen.
Unsere Forschung hat gezeigt, dass bakterielle Erreger bei der Infektion essenziell auf die Kooperation von Wirtsproteinen angewiesen sind. Aus diesen Erkenntnissen könnten sich neue Strategien zur Infektionsbekämpfung ergeben.
Unsere Forschung
Die aktuellen Berichte der Weltgesundheitsorganisation (WHO) mit dem Titel „The Global Burden of Disease“ (GBD) belegen deutlich die Wichtigkeit der Forschung an Wirt-Pathogen-Interaktionen. Evolution ist ein kontinuierlicher Prozess, der immer stattfindet und hochvirulente und multiresistente Bakterienstämme wie auch sogenannte „emerging pathogens“ hervorbringt. Ein tiefes Verständnis für das komplexe Wechselspiel zwischen krankheitserregenden Bakterien und dem Wirt, ist unabdingbar, um diesen Problemen in Zukunft angemessen begegnen zu können.
Kernexpertise: Kombination von Videomikroskopie und Proteinbiochemie
Die HZI-Abteilung Zellbiologie befasst sich sowohl mit den Verteidigungsmechanismen des Wirtes als auch mit den Virulenzmechanismen der Pathogene. Die Kernexpertise liegt in der Kombination aus Videomikroskopie und Proteinbiochemie, angewendet an verschiedensten Zellbewegungsmodellen und im Kontext bakterieller Infektion. In der Vergangenheit konnte die Abteilung sowohl grundlegende Regulationsmechanismen der Zellbewegung aufklären, als auch Beispiele für deren Beeinflussung durch bakterielle Virulenzfaktoren aufzeigen.
Unsere Forschung hat gezeigt, dass bakterielle Erreger bei der Infektion essenziell auf die Kooperation von Wirtsproteinen angewiesen sind. Aus diesen Erkenntnissen könnten sich neue Strategien zur Infektionsbekämpfung ergeben.
Prof. Dr. Theresia Stradal
Das Wechselspiel zwischen krankheitserregenden Bakterien und Wirtszellen ist höchst komplex und nur unzureichend verstanden. Ein besseres Verständnis dieser Schnittstelle zwischen Molekülen beider Welten, Bakterien und Wirtszellen, könnte neue Ansätze liefern, um die oft fatale Interaktion zu unterbinden und den Weg für neue anti-infektive Strategien zu ebnen.

Theresia Stradal studierte Biologie an der Paris-Lodron-Universität zu Salzburg in Österreich, an der sie im Jahr 2000 am Institut für Molekularbiologie promovierte. Anschließend arbeitete sie für vier Jahre als Postdoc in der Abteilung Zellbiologie bei der Gesellschaft für Biotechnologische Forschung (heute: Helmholtz-Zentrum für Infektionsforschung, HZI) in Braunschweig.
Ab 2005 war sie dort als Forschungsgruppenleiterin tätig, bis sie 2009 von der Westfälischen Wilhelms Universität Münster zur Professorin (W2) berufen wurde und zum dortigen Institut für Molekulare Zellbiologie wechselte. 2014 kehrte sie nach Braunschweig zurück, wo sie eine W3-Professur am Institut für Zoologie der Technischen Universität Braunschweig annahm.
Zeitgleich wurde Theresia Stradal Leiterin der Abteilung Zellbiologie am HZI. Hier forscht sie im Bereich der Wirt-Pathogen-Interaktionen und untersucht die Verteidigungsmechanismen des Wirtes sowie die Virulenzmechanismen der Pathogene.
Team








Ausgewählte Publikationen
Stradal TE, Courtney KD, Rottner K, Hahne P, Small JV, Pendergast AM (2001) The Abl interactor proteins localize to sites of actin polymerization at the tips of lamellipodia and filopodia. Curr Biol 11: 891-895.
Steffen A, Rottner K, Ehinger J, Innocenti M, Scita G, Wehland J, Stradal TE (2004) Sra-1 and Nap1 link Rac to actin assembly driving lamellipodia formation. EMBO J 23: 749-759.
Disanza A, Mantoani S, Hertzog M, Gerboth S, Frittoli E, Steffen A, Berhoerster K, Kreienkamp HJ, Milanesi F, Di Fiore PP, Ciliberto A, Stradal TE, Scita G (2006) Regulation of cell shape by Cdc42 is mediated by the synergic actin bundling activity of the Eps8:IRSp53 complex. Nat Cell Biol 8: 1337-1347.
Weiss SM, Ladwein M, Schmidt D, Ehinger J, Lommel S, Städing K, Beutling U, Disanza A, Frank R, Jänsch L, Scita G, Gunzer F, Rottner K, Stradal TE (2009) IRSp53 links Tir to EspFU/N-WASP-mediated actin assembly in EHEC pedestal formation. Cell Host Microbe 5: 244-258.
Hänisch J, Kölm R, Wozniczka M, Bumann D, Rottner K, Stradal TE (2011) Activation of a RhoA/myosin II-dependent but Arp2/3 complex-independent pathway facilitates Salmonella invasion. Cell Host Microbe 9: 273-285.
Publikationen
Review articles
Stradal TE, Costa SC. (2017) Type III Secreted Virulence Factors Manipulating Signaling to Actin Dynamics. Curr Top Microbiol Immunol. 399:175-199. doi: 10.1007/82_2016_35.
Rottner K, Stradal TE. (2016) How distinct Arp2/3 complex variants regulate actin filament assembly. Nat Cell Biol.;18(1):1-3.
Rottner, K., Stradal, T.E. (2011) Actin dynamics and turnover in cell motility. Curr. Opin. Cell. Biol. 23(5), 569-578.
Rottner, K., Stradal, T.E. (2009) Poxviruses taking a ride on actin: new users of known hardware. Cell Host & Microbe 6(6), 497-499.
Faix, J., Breitsprecher, D., Stradal, T.E., Rottner, K. (2009) Filopodia: Complex models for simple rods. Int. J. Biochem. Cell Biol. 41(8-9), 1656-1664.
Stradal, T.E., Scita, G. (2006) Protein complexes regulating cellular actin assembly. Curr. Op. Cell Biol. 18, 4-10.
Rottner, K., Stradal, T.E., Wehland, J. (2005) Bacteria – host cell interactions at the plasma membrane: stories on actin cytoskeleton subversion. Dev. Cell 9, 3-17.
Rottner, K., Lommel, S., Wehland, J., Stradal, T.E. (2004) Pathogen-induced actin filament rearrangement in infectious diseases. J. Pathol. 204, 396-406.
Stradal, T.E., Rottner, K., Disanza, A., Innocenti, M., Confalonieri, S., Scita, G. (2004). Regulation of actin dynamics by WASP and WAVE family proteins. Trends Cell Biol. 14, 303-311.
Small, J.V., Stradal, T.E., Vignal, E., Rottner, K. (2002) The lamellipodium: where motility begins. Trends Cell Biol. 12, 112-120.
Stradal, T.E., Kranewitter, W., Winder, S.J., Gimona, M. (1998) CH domains revisited. FEBS Lett. 431, 134-137.
Book contributions
Steffen, A., Stradal, T.E., Rottner, K. (2016) Signalling pathways controlling cellular actin organization. The Actin Cytoskelton in “Handbook of Experimental Pharmacology” (ed. B. Jockusch) Springer Books (in press)
Stradal, T.E., Pusch, R., Kliche, S. (2006) Molecular regulation of cytoskeletal rearrangements during T cell signalling. In: Results and Problems in Cell Differentiation (RPCD); Volume: Cell Communication in Nervous and Immune System (eds. B. Schraven & E. Gundelfinger), Springer Verlag, Berlin, 43, pp219-244.
Rottner, K., Kaverina, I.N., Stradal, T.E. (2006) Cytoskeleton proteins. In “Cell Biology: A Laboratory Handbook”, 3rd Edition (ed. J.E. Celis), Academic Press, pp111-119.
Stradal, T.E., Lommel, S., Wehland, J., Rottner, K. (2005) Host-pathogen interactions and Cell Motility: Learning from Bacteria. In “Cell Migration in Development and Disease” (ed. D. Wedlich), WILEY-VCH Verlag GmbH & Co. KGaA, Weinheim, pp205-248.
Stradal, T.E., Sechi, A.S., Wehland, J., Rottner, K. (2003) The Cytoskeleton. In “Essential Cell Biology, Vol 1, ‘Cell Structure’: Practical Approach Series” (eds. M. Davey & M. Lord), Oxford University Press, pp365-389.
Other
Stradal, T.E., Backert, S. (2010) Host-pathogen interaction: How EHEC influence the actin cytoskeleton of the host cell. BioSpektrum 16 (6), 624-627.
Stradal, T.E. (2008) The regulation of cellular protrusions. Zellbiologie Aktuell 34(1), 17-22.
Stradal, T.E., Wehland, J. (2005) Aktindynamik und WASP/WAVE-Proteine. BioSpektrum 11(3), 283-286.
Aktuelle Projekte
Role of the WAVE complex in the immune system
Actin branching at the cell periphery depends on WAVE- and Arp2/3-complexes. Our group has a long history in WAVE-complex research and contributed significantly to the elucidation of its function. Over the years, we analyzed tissue-cultured cells suppressed in the expression of its subunits by RNA-interference or genetic deletion. To unambiguously clarify the role of WAVE-complex and thus lamellipodia in vivo, we decided to target WAVE-complex in the immune system, where motile cells fulfil defined functions while showing different types of motile behavior. Deletion of Hem1 results in the loss of WAVE-complex and consequently lamellipodium formation in all immune cells. Strikingly, Hem1 deletion eliminates chemotaxis. In complex 3D-environments, lamellipodia mediate exploration of the environment. Consequently, Hem1-null cells can no longer make directional decisions, persistently crawl in one direction and thus likely become trapped in narrow pores. These trapped yet activated innate immune cells then cause local sterile inflammation. Dendritic cells derived from these mice show abberant migratory behavior and fail to perform interstitial migration and chemotaxis (Leithner et al., 2016, Thiam et al., 2016).
Geldgeber/Förderer:
DFG - Deutsche Forschungsgemeinschaft
Relevant publications concerning this project
I. Leithner A, Eichner A, Müller J, Reversat A, Brown M, Schwarz J, Merrin J, de Gorter DJ, Schur F, Bayerl J, de Vries I, Wieser S, Hauschild R, Lai FP, Moser M, Kerjaschki D, Rottner K, Small JV, Stradal TE, Sixt M (2017) Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nat Cell Biol 18(11): 1253-59.
II. Thiam HR, Vargas P, Carpi N, Crespo CL, Raab M, Terriac E, King MC, Jacobelli J, Alberts AS, Stradal T, Lennon-Dumenil AM, Piel M (2016) Perinuclear Arp2/3-driven actin polymerization enables nuclear deformation to facilitate cell migration through complex environments. Nat Commun 7: 10997.
III. Kage F, Winterhoff M, Dimchev V, Mueller J, Thalheim T, Freise A, Brühmann S, Kollasser J, Block J, Dimchev G, Geyer M, Schnittler HJ, Brakebusch C, Stradal TE, Carlier MF, Sixt M, Käs J, Faix J, Rottner K (2017) FMNL formins boost lamellipodial force generation. Nat Commun 8:14832.
IV. Rottner K, Stradal TE (2016) How distinct Arp2/3 complex variants regulate actin filament assembly. Nat Cell Biol 18(1): 1-3.
V. Litschko C, Linkner J, Brühmann S, Stradal TEB, Reinl T, Jänsch L, Rottner K, Faix J (2017) Differential functions of WAVE regulatory complex subunits in the regulation of actin-driven processes. Eur J Cell Biol 96(8): 715-27.
VI. Steffen A, Ladwein M, Dimchev GA, Hein A, Schwenkmezger L, Arens S, Ladwein KI, Margit Holleboom J, Schur F, Victor Small J, Schwarz J, Gerhard R, Faix J, Stradal TE, Brakebusch C, Rottner K (2013) Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. J Cell Sci 126(Pt 20): 4572-88.
VII. Tahirovic S, Hellal F, Neukirchen D, Hindges R, Garvalov BK, Flynn KC, Stradal TE, Chrostek-Grashoff A, Brakebusch C, Bradke F (2010) Rac1 regulates neuronal polarization through the WAVE complex. J Neurosci 30(20): 6930-43.
VIII. Derivery E, Fink J, Martin D, Houdusse A, Piel M, Stradal TE, Louvard D, Gautreau A (2008) Free Brick1 is a trimeric precursor in the assembly of a functional wave complex. PLoS ONE 3(6): e2462.
IX. Steffen A, Faix J, Resch GP, Linkner J, Wehland J, Small JV, Rottner K, Stradal TE (2006) Filopodia formation in the absence of functional WAVE- and Arp2/3-complexes. Mol Biol Cell 17(6): 2581-91.
X. Stradal TE, Scita G (2006) Protein complexes regulating Arp2/3-mediated actin assembly. Curr Opin Cell Biol 18(1): 4-10.
XI. Innocenti M, Gerboth S, Rottner K, Lai FP, Hertzog M, Stradal TE, Frittoli E, Didry D, Polo S, Disanza A, Benesch S, Di Fiore PP, Carlier MF, Scita G (2005) Abi1 regulates the activity of N-WASP and WAVE in distinct actin-based processes. Nat Cell Biol 7(10): 969-76.
XII. Innocenti M, Zucconi A, Disanza A, Frittoli E, Areces LB, Steffen A, Stradal TE, Di Fiore PP, Carlier MF, Scita G (2004) Abi1 is essential for the formation and activation of a WAVE2 signalling complex. Nat Cell Biol 6(4): 319-27.
XIII. Steffen A, Rottner K, Ehinger J, Innocenti M, Scita G, Wehland J, Stradal TE (2004) Sra-1 and Nap1 link Rac to actin assembly driving lamellipodia formation. EMBO J 23(4): 749-59.
XIV. Kaverina I, Stradal TE, Gimona M (2003) Podosome formation in cultured A7r5 vascular smooth muscle cells requires Arp2/3-dependent de-novo actin polymerization at discrete microdomains. J Cell Sci 116(Pt 24): 4915-24.
XV. Small JV, Stradal T, Vignal E, Rottner K (2002) The lamellipodium: where motility begins. Trends Cell Biol 12(3): 112-20.
XVI. Stradal T, Courtney KD, Rottner K, Hahne P, Small JV, Pendergast AM (2001) The Abl interactor proteins localize to sites of actin polymerization at the tips of lamellipodia and filopodia. Curr Biol 11(11): 891-5.
Technologieangebote
Folgende Technologien wurden von der Abteilung "Zellbiologie" entwickelt und zum Patent angemeldet:
Efficient Generation of Murine Brain Organoids from Adult Neural Stem Cells