Antivirale und Antivirulenzwirkstoffe

Unsere Forschung
Wir verfolgen unsere Projekte durch alle Phasen der Wirkstoffentdeckung. Unsere Strategien zur Auffindung von Hitverbindungen sind individuell auf das Zielprotein zugeschnitten. Im Falle von Rezeptoren und Enzymen mit tiefen Taschen, welche mit niedermolekularen Verbindungen leicht addressiert werden können, verwenden wir üblicherweise Fragment-basierte Methoden und optimieren die so identifizierten Startpunkte (Hits) hin zu verbesserten Leitstrukturen (Leads) durch medizinalchemische Optimierung. Dabei verfolgen wir einen multi-Parameter-Ansatz, welcher zusätzlich zu Potenz auch pharmakokinetische sowie sicherheitspharmakologische Aspekte berücksichtigt. Idealerweise, können die Leitstrukturen dann zu präklinischen Profilierungskandidaten für die (prä-)klinische Entwicklung weiteroptimiert werden.
Makromolekül-makromolekül-Interaktionen sind schwierig mit niedermolekularen Verbindungen zu adressieren. Dennoch sind dies hoch interessante und wenig explorierte Zielpunkte für Wirkstofforschung. Wir waren darin erfolgreich die oben genannte Fragment-basierte Hitidentifikationsstrategie auch auf eine Protein-DNA-Interaktion anzuwenden, welche essentiell für den Lebenszyklus des humanen Herpesvirus 8 ist (Kaposi-Sarkom-Herpesvirus, KSHV). Diese Interaktion neeinhaltet das sog. Latency-associated nuclear antigen (LANA). Mit dem Ziel nun diesen niedermolekularen Ansatz zu komplementieren, wenden wir die Phagen-Display Technologie an um sehr kurze makrozyklische und konformationell eingeschränkte Peptide zu finden. Die resultierenden Hits sind sehr gut synthetisch zugänglich und bieten ideale Startpunkte für eine medizinalchemische Optimierung hin zu neuen Wirkstoffmolekülen an den Grenzen der Lipinski-Regeln (bzw. leicht jenseits dessen). Zukünftig wollen wir die Fragment- und Phagen-basierten Ansätze miteinander vereinen, um Peptid-Fragment-Hybridmoleküle zu generieren und dadurch in bisher ungenutzten chemischen Strukturraum vorzudringen.
Unsere Forschung
Wir verfolgen unsere Projekte durch alle Phasen der Wirkstoffentdeckung. Unsere Strategien zur Auffindung von Hitverbindungen sind individuell auf das Zielprotein zugeschnitten. Im Falle von Rezeptoren und Enzymen mit tiefen Taschen, welche mit niedermolekularen Verbindungen leicht addressiert werden können, verwenden wir üblicherweise Fragment-basierte Methoden und optimieren die so identifizierten Startpunkte (Hits) hin zu verbesserten Leitstrukturen (Leads) durch medizinalchemische Optimierung. Dabei verfolgen wir einen multi-Parameter-Ansatz, welcher zusätzlich zu Potenz auch pharmakokinetische sowie sicherheitspharmakologische Aspekte berücksichtigt. Idealerweise, können die Leitstrukturen dann zu präklinischen Profilierungskandidaten für die (prä-)klinische Entwicklung weiteroptimiert werden.
Makromolekül-makromolekül-Interaktionen sind schwierig mit niedermolekularen Verbindungen zu adressieren. Dennoch sind dies hoch interessante und wenig explorierte Zielpunkte für Wirkstofforschung. Wir waren darin erfolgreich die oben genannte Fragment-basierte Hitidentifikationsstrategie auch auf eine Protein-DNA-Interaktion anzuwenden, welche essentiell für den Lebenszyklus des humanen Herpesvirus 8 ist (Kaposi-Sarkom-Herpesvirus, KSHV). Diese Interaktion neeinhaltet das sog. Latency-associated nuclear antigen (LANA). Mit dem Ziel nun diesen niedermolekularen Ansatz zu komplementieren, wenden wir die Phagen-Display Technologie an um sehr kurze makrozyklische und konformationell eingeschränkte Peptide zu finden. Die resultierenden Hits sind sehr gut synthetisch zugänglich und bieten ideale Startpunkte für eine medizinalchemische Optimierung hin zu neuen Wirkstoffmolekülen an den Grenzen der Lipinski-Regeln (bzw. leicht jenseits dessen). Zukünftig wollen wir die Fragment- und Phagen-basierten Ansätze miteinander vereinen, um Peptid-Fragment-Hybridmoleküle zu generieren und dadurch in bisher ungenutzten chemischen Strukturraum vorzudringen.
Prof. Dr. Martin Empting
Als akademische Wirkstoffforscher haben wir ein klares Mandat die Innovation im Bereich der Anti-Infektivaforschung voranzutreiben. Dies erreichen wir unter anderem durch Erschließung und Ausnutzung neuer Wirkweisen (Modes-of-Action) zur Behandlung viraler und bakterieller Infektionen durch Methoden der Medizinischen Chemie.

Dr. Martin Empting studierte Chemie an der Technischen Universität Darmstadt. Für seinen Diplomabschluss erhielt er den Preis der „Dr. Anton-Keller-Stiftung“. Danach im Zuge seiner Doktorarbeit unter der Leitung von Prof. Dr. Harald Kolmar und fokussierte sind in dieser Zeit fokussierte auf peptidische Wirkstoffkandidaten und biomimetische Konzepte. Seine Dissertation, welche er 2013 mit summa cum laude abschloss, wurde mit dem Preis der „Familie Bottling-Stiftung“ ausgezeichnet. Im Zuge eines PostDoc unter der Leitung von Prof. Dr. Rolf W. Hartmann am Helmholtz Institut für Pharmazeutische Forschung Saarland (HIPS) richtete er seinen Fokus auf das Design und die Optimierung von neuen Antiinfektiva gegen Gramnegative Bakterien. Seit 2017, verfolgt er seine eigenständige Forschung mit dem Ziel neue synthetische Wirkstoffmoleküle zu generieren, die für eine Entwicklung hin zu dringend benötigten antiinfektiven Arzneimitteln geeignet sind.
Ausgewählte Publikationen
A. Berwanger, S. C. Stein, A. M. Kany, M. Gartner, B. Loretz, C.-M. Lehr, A. K. H. Hirsch, T. F. Schulz, M. Empting, Disrupting Kaposi’s Sarcoma-Associated Herpesvirus (KSHV) latent replication with a small molecule inhibitor, Journal of Medicinal Chemistry (2023), 66, 10782-10790, https://doi.org/10.1021/acs.jmedchem.3c00990.
M. M. Hamed, A. S. Abdelsamie, K. Rox, C. Schütz, A. M. Kany, T. Röhrig, S. Schmelz, W. Blankenfeldt, A. Arce-Rodriguez, J. Manuel Borrero-de Acuña, D. Jahn, J. Rademacher, F. Ringshausen, B. Tümmler, A. K. H. Hirsch, R. W. Hartmann, M. Empting, Towards translation of PqsR inverse agonists: from in vitro efficacy optimization to in vivo proof-of-principle, Advanced Science (2023), 10, 2204443, https://doi.org/10.1002/advs.202204443.
V. Jakob, B. G. E. Zoller, J. Rinkes, Y. Wu, A. F. Kiefer, M. Hust, S. Polten, A. M. White, P. J. Harvey, T. Durek, D. J. Craik, A. Siebert, U. Kazmaier, M. Empting, Phage display-based discovery of cyclic peptides against the broad spectrum bacterial anti-virulence target CsrA, European Journal of Medicinal Chemistry (2022), 231, 114148, https://doi.org/10.1016/j.ejmech.2022.114148
C. Schütz, D.-K. Ho, M. Hamed, A. S. Abdelsamie, T. Röhrig, C. Herr, A. M. Kany, K. Rox, S. Schmelz, L. Siebenbürger, M. Wirth, C. Börger, S. Yahiaoui, R. Bals, A. Scrima, W. Blankenfeldt, J. C. Horstmann, R. Christmann, X. Murgia, M. Koch, A. Berwanger, B. Loretz, A. K. H. Hirsch, R. W. Hartmann, C.-M. Lehr, M. Empting, A new PqsR inverse agonist potentiates tobramycin efficacy to eradicate Pseudomonas aeruginosa biofilms, Advanced Science (2021), 8, 2004369, https://doi.org/10.1002/advs.202004369
P. Kirsch, V. Jakob, K. Oberhausen, S.C. Stein, I. Cucarro, T.F. Schulz, M. Empting, Fragment-based discovery of a qualified hit targeting the latency-associated nuclear antigen of the oncogenic Kaposi's sarcoma-associated herpesvirus/human herpesvirus 8, Journal of Medicinal Chemistry (2019), 62, 3924-3939, https://doi.org/10.1021/acs.jmedchem.8b01827
Eine komplette Liste aller Publikationen finden Sie auf der HIPS-Webseite.
Technologieangebote
Folgende Technologien wurden von der Abteilung Antivirale und Antivirulenzwirkstoffe entwickelt und zum Patent angemeldet:
Projekte
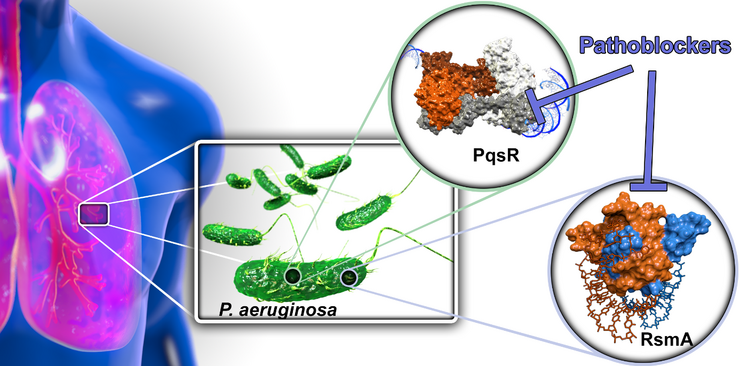
Bakterielle Pathogenität aushebeln (Pathoblocker)
Mit dem Ziel neue Behandlungsmodalitäten für bakterielle Infektionen zu finden, untersuchen wir Möglichkeiten die klassischen Wirkweisen von bakteriziden bzw. bakteriostatischen Antiotika zu umgehen. Dazu designen und optimieren wir synthetische Moleküle, welche spezifisch bakterielle Pathogenitätseigenschaften blockieren. Um dieses zu erreichen adressieren wir sowohl Transkriptionregulatoren als auch post-transkriptionale Regulatoren der bakteriellen Virulenz. Dadurch sind wir in der Lage mit einer Reihe von Virulenzfaktoren auszuschalten ohne mit der Lebensfähigkeit der Bakterien zu interferieren. Hierbei wollen wir gezielt Verbindungen für die Behandlung von Infektionen durch Pseudomonas aeruginosa finden und voranbringen, welches ein gefährliches ESKAPE Pathogen und die Hauptursache für Morbidität und Mortalität bei Mukoviszidose ist. Hierbei adressieren wir zwei Regulatorsysteme: A) das Pseudomonas quinolone signal (PQS) Quorum Sensing (QS) System und B) das carbon storage regulator (Csr; auch regulator of secondary metabolites, Rsm) System.
A) QS ist ein Weg für Bakterien untereinander mittels kleiner diffundierender Signalmoleküle zu kommunizieren. Diese Eigenschaft ist essentiell für die Koordinierung von populationsweiten Verhaltensanpassungen im Infektionsprozess. Wir adressieren den Signalmolekülrezeptor PqsR (MvfR) durch sog. Inverse Agonisten. Das PQS QS System ist unter Bakterien recht einzigartig weswegen unsere Verbindungen Pathogen-spezifisch wirken und somit das kommensale (gutartige) Mirkobiom nicht angreifen. Wir haben bereits erfolgreich Leitstrukturstatus mit drei chemischen Klassen erreicht. Diese Pathoblocker besitzen hohe Potenz im nanomolaren Bereich sowie geeignete pharmakokinetische Eigenschaften und vielversprechende sicherheitspharmakologische Profile. Dieses Projekt befindet sich derzeit in der späten Leitstrukturoptimierungsphase.
B) Im Gegensatz zum PQS QS System ist das Csr/Rsm System unter bakteriellen Pathogenen erstaunlich weit verbreitet. Daher könnte dieses höher rangige Regulationssystem die Möglichkeit eröffnen breit-band Pathoblocker zu generieren. Wir waren bereits darin erfolgreich, sowohl Naturstoff-abgeleitete niedermolekulare Hits als auch neuartige peptidische Makrozyklen als Startpunkte für die Medizinalchemie zu identifizieren. Ein wichtiger nächster Schritt wird die Demonstration von zellulären Effekten durch die Blockade dieses post-transkriptionalen Regulators.
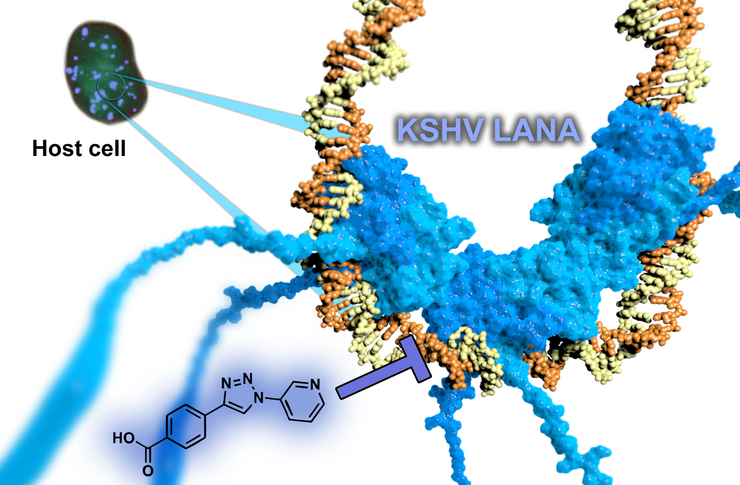
Aufhebung der Persistenz von humanen Herpesviren
Das virale Protein latency-associated nuclear antigen (LANA) ist essentiell für die latente Replikation und Persistenz des humanen Herpesvirus 8/Kaposi-Sarkom-Herpesvirus. Dabei übernimmt LANA wichtige Funktionen für das Überleben des Virus in der Wirtszelle wie Transkriptionskontrolle, latente Replikation der viralen DNA und Episomsegregation während der Mitose. Es übt Teile seiner Funktionen durch die Anknüpfung des viralen Episomes an das Wirtschromatin. Daher ist es unser Ziel, neue antivirale Wirkstoffe zu finden, welche die Interaktion zwischen LANA und dem viralen Genom inhibieren. Dadurch sollte es möglich sein die latente Replikation von KSHV zu unterbinden.
Wir waren bereits erfolgreich in der Identifizierung der allerersten LANA-Inhibitoren durch einen Fragment-basierten Ansatz. Der resultierende optimierte Hit stellt aufgrund der nachgewiesenen Bindung an das Target, einer hohen Ligandeneffizienz und guten physikochemischen Eigenschaften einen idealen Startpunkt für die nun laufende Hit-to-Lead Kampagne dar mit dem Ziel antivirale Aktivität in Zell-basierten Assays zu zeigen.