Bakterielle Infektionsökologie

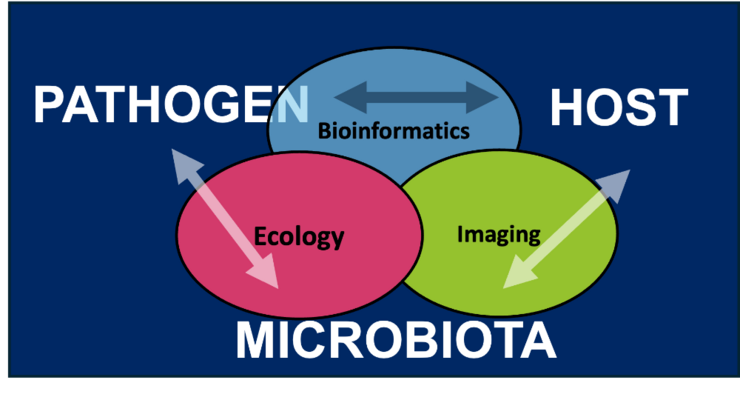
Unser Körper beherbergt vielfältige mikrobielle Gemeinschaften, die uns vor Krankheitserregern schützen, wobei unsere Schleimhäute einen besonders dicht besiedelten Lebensraum darstellen. Wir streben danach, die Mechanismen zu bestimmen, die die Interaktion zwischen Mikrobiota, Krankheitserregern und Wirtszellen steuern. Mit einem multidisziplinären Ansatz, der ökologische Theorie, Bioinformatik und neue bildgebende Verfahren kombiniert, konzentriert sich unsere Forschung auf drei Schlüsselthemen:
Kernprinzipien der räumlichen Organisation des Mikrobioms
Mikrobielle Gemeinschaften bestehen aus vielen Arten, und ihre räumliche Anordnung auf Einzelzellebene bestimmt, ob sie sich tatsächlich treffen und interagieren. Aktuelle Methoden sind oft nicht in der Lage, Interaktionen auf dieser feinen Ebene in Wirten zu erfassen, was zu einer Auflösungslücke genau dort führt, wo wichtige ökologische und evolutionäre Prozesse ablaufen. Mit der Entwicklung neuer Imaging-Technologien und definierten räumlichen Modellen schließen wir diese Lücke und erfassen die räumliche Ökologie von Mikrobiomen auf zellulärer Ebene. Dabei vergleichen wir systematisch gesunde und kranke Zustände. Diese Daten liefern ein Verständnis über die strukturellen Organisationsprinzipien unserer Mikrobiota und wie die Resistenz gegen Störungen wie Antibiotika, Entzündungen und Infektionen gestärkt werden kann.
Einfluss der Mikrobiota auf Infektionen

Wie verhindern unsere bakteriellen Gemeinschaften Infektionen? In diesem Kontext untersuchen wir die grundlegenden Mechanismen der Mikrobiom-vermittelten Pathogenresistenz, insbesondere bei intestinalen Infektionen und damit verbundenen Entzündungen. Dabei untersuchen wir, wie die räumlichen Interaktionen zwischen Mikrobiota und Pathogenen den Verlauf von Infektionen beeinflussen. Ziel ist es, Schlüsselmikrobiota zu identifizieren, die zur Infektionsresistenz beitragen, und die zugrunde liegende Ökologie von Infektionen im Wirt aufzuklären. Dies wird weiterhin die Neuformulierung von mikrobiellen Gemeinschaften auf Grundlage ihrer Biogeographie ermöglichen, um die genaue Rolle dieser Taxa bei Infektionen zu entschlüsseln. Im Ergebnis soll diese Forschung neue Erkenntnisse für das rationale Design nützlicher mikrobieller Antiinfektiva liefern.
Wirtsmanipulation und mikrobiom-inspirierte Therapeutika
Wie manipulieren Bakterien ihren Wirt? Dazu screenen wir humanassoziierte Bakterien mit einer Kombination aus Bioinformatik und Zellassays. Erkenntnisse über mikrobiom-vermittelte Wirtsmanipulation bieten hier ein großes Potenzial, von der Natur zu lernen und Kandidaten für gezielte Anwendungen zu finden, wie z.B. bei chronisch-entzündlichen Erkrankungen (z.B. IBD). Darüber hinaus eröffnen sich Anwendungsmöglichkeiten durch das Verständnis der Mikrobiom-vermittelten Immunmodulation im Verlauf von Infektionen.
Unser Körper beherbergt vielfältige mikrobielle Gemeinschaften, die uns vor Krankheitserregern schützen, wobei unsere Schleimhäute einen besonders dicht besiedelten Lebensraum darstellen. Wir streben danach, die Mechanismen zu bestimmen, die die Interaktion zwischen Mikrobiota, Krankheitserregern und Wirtszellen steuern. Mit einem multidisziplinären Ansatz, der ökologische Theorie, Bioinformatik und neue bildgebende Verfahren kombiniert, konzentriert sich unsere Forschung auf drei Schlüsselthemen:
Kernprinzipien der räumlichen Organisation des Mikrobioms

Mikrobielle Gemeinschaften bestehen aus vielen Arten, und ihre räumliche Anordnung auf Einzelzellebene bestimmt, ob sie sich tatsächlich treffen und interagieren. Aktuelle Methoden sind oft nicht in der Lage, Interaktionen auf dieser feinen Ebene in Wirten zu erfassen, was zu einer Auflösungslücke genau dort führt, wo wichtige ökologische und evolutionäre Prozesse ablaufen. Mit der Entwicklung neuer Imaging-Technologien und definierten räumlichen Modellen schließen wir diese Lücke und erfassen die räumliche Ökologie von Mikrobiomen auf zellulärer Ebene. Dabei vergleichen wir systematisch gesunde und kranke Zustände. Diese Daten liefern ein Verständnis über die strukturellen Organisationsprinzipien unserer Mikrobiota und wie die Resistenz gegen Störungen wie Antibiotika, Entzündungen und Infektionen gestärkt werden kann.
Einfluss der Mikrobiota auf Infektionen

Wie verhindern unsere bakteriellen Gemeinschaften Infektionen? In diesem Kontext untersuchen wir die grundlegenden Mechanismen der Mikrobiom-vermittelten Pathogenresistenz, insbesondere bei intestinalen Infektionen und damit verbundenen Entzündungen. Dabei untersuchen wir, wie die räumlichen Interaktionen zwischen Mikrobiota und Pathogenen den Verlauf von Infektionen beeinflussen. Ziel ist es, Schlüsselmikrobiota zu identifizieren, die zur Infektionsresistenz beitragen, und die zugrunde liegende Ökologie von Infektionen im Wirt aufzuklären. Dies wird weiterhin die Neuformulierung von mikrobiellen Gemeinschaften auf Grundlage ihrer Biogeographie ermöglichen, um die genaue Rolle dieser Taxa bei Infektionen zu entschlüsseln. Im Ergebnis soll diese Forschung neue Erkenntnisse für das rationale Design nützlicher mikrobieller Antiinfektiva liefern.
Wirtsmanipulation und mikrobiom-inspirierte Therapeutika
Wie manipulieren Bakterien ihren Wirt? Dazu screenen wir humanassoziierte Bakterien mit einer Kombination aus Bioinformatik und Zellassays. Erkenntnisse über mikrobiom-vermittelte Wirtsmanipulation bieten hier ein großes Potenzial, von der Natur zu lernen und Kandidaten für gezielte Anwendungen zu finden, wie z.B. bei chronisch-entzündlichen Erkrankungen (z.B. IBD). Darüber hinaus eröffnen sich Anwendungsmöglichkeiten durch das Verständnis der Mikrobiom-vermittelten Immunmodulation im Verlauf von Infektionen.
Das Verständnis der Interaktionen zwischen uns, unseren Mikroben und Krankheitserregern wird innovative Therapien hervorbringen, die unsere Gesundheit und unser Wohlbefinden verbessern.

Martin Jahn ist Mikrobiologe, Ökologe und Bioinformatiker der erforscht, wie Mikroben mit unserem Körper interagieren und unsere Gesundheit beeinflussen. Er studierte Naturwissenschaften mit den Schwerpunkten Mikrobiologie, Ökologie und Bioinformatik an der Universität Würzburg. Während dieser Zeit führte er als Gastwissenschaftler an der Universität Glasgow mit Prof. Roman Biek Feldforschungen zur räumlichen Ökologie von Borrelia burgdorferi in Wirtsreservoiren durch.
Nach dem Studium erhielt Martin Jahn ein Stipendium zur Promotion an der Universität Kiel im Sonderforschungsbereich 1182 bei Prof. Ute Hentschel. Dort untersuchte er die funktionellen Grundlagen von Schwamm-Symbiosen, der ältesten bekannten Tier-Mikroben-Symbiose. Im Jahr 2018 führte ihn seine Arbeit als Gastwissenschaftler an die Universität Lausanne, wo er mit Prof. Anders Meibom an korrelativer metabolischer Bildgebung arbeitete.
2019 promovierte Martin Jahn mit summa cum laude und erhielt 2020 ein Stipendium des Human Frontier Science Programme (HFSP), um seine wissenschaftliche Ausbildung an der Universität Oxford fortzusetzen. In Oxford arbeitete er als Postdoktorand bei Professor Kevin Foster und beschäftigte sich mit der Biogeographie des Darmmikrobioms und dessen Auswirkungen auf die Krankheitsresistenz. Dies beinhaltete die Entwicklung neuer Technologien zur Kartierung der zellulären Organisation ganzer wirtsassoziierter Bakteriengemeinschaften. Der Schwerpunkt lag dabei auf strukturellen Veränderungen des Mikrobioms in verschiedenen Krankheitskontexten. Diese Erfahrung prägte seine Sichtweise auf den menschlichen Körper als komplexes und vielfältiges Ökosystem, in dem mikrobielle Interaktionen eine entscheidende Rolle bei der Erhaltung der Gesundheit spielen.
Seit Oktober 2024 ist Martin Jahn Gruppenleiter am HZI Braunschweig, wo er die Gruppe Bakterielle Infektionsökologie leitet.
Team



Ausgewählte Publikationen
Jahn M.T., Arkhipova K. Markert S.M., Stigloher C., Lachnit T., Pita L., Kupczok A., Ribes M., Stengel S.T., Rosenstiel P., Dutilh B.E., Hentschel U. (2019). A Phage Protein Aids Bacterial Symbionts in Eukaryote Immune Evasion. Cell Host & Microbe; doi: 10.1016/j.chom.2019.08.019
Spragge F, Bakkeren E, Jahn, M.T., Araujo EBN, Pearson CF, Wang X, Pankhurst L, Cunrath O, Foster KR 2023 Microbiome diversity protects against pathogens by nutrient blocking. Science; doi: 10.1126/science.adj3502
Jahn, M.T., Lachnit, T., Markert, S.M., C. Stigloher, L. Pita, M. Ribes, B. E. Dutilh,. U. Hentschel (2021). Lifestyle of sponge symbiont phages by host prediction and correlative microscopy. ISME J; doi: 10.1038/s41396-021-00900-6
Jahn M.T., Markert S.M., Ryu T, Ravasi T, Stigloher C, Hentschel U, Moitinho-Silva (2016) Shedding light on cell compartmentation in the candidate phylum Poribacteria by high resolution visualisation and transcriptional profiling. Sci. Rep. 6; doi: 10.1038/srep35860
Hildebrand F, Moitinho-Silva L, Blasche S, Jahn M.T., Gossmann T.I., Heuerta-Cepas J, Hercog R, Luetge M, Bahram M, Pryszlak A, Alves R.J., Waszak S.M., Zhu A, Ye L, Costea P.I., Aalvink S, Belzer C, Forslund S.K., Sunagawa S, Hentschel U, Merten C, Patil K.R., Benes V, Bork P (2019). Antibiotics-induced monodominance of a novel gut bacterial order. Gut; doi: 10.1136/gutjnl-2018-317715
