Dynamik respiratorischer Infektionen

Unsere Forschung
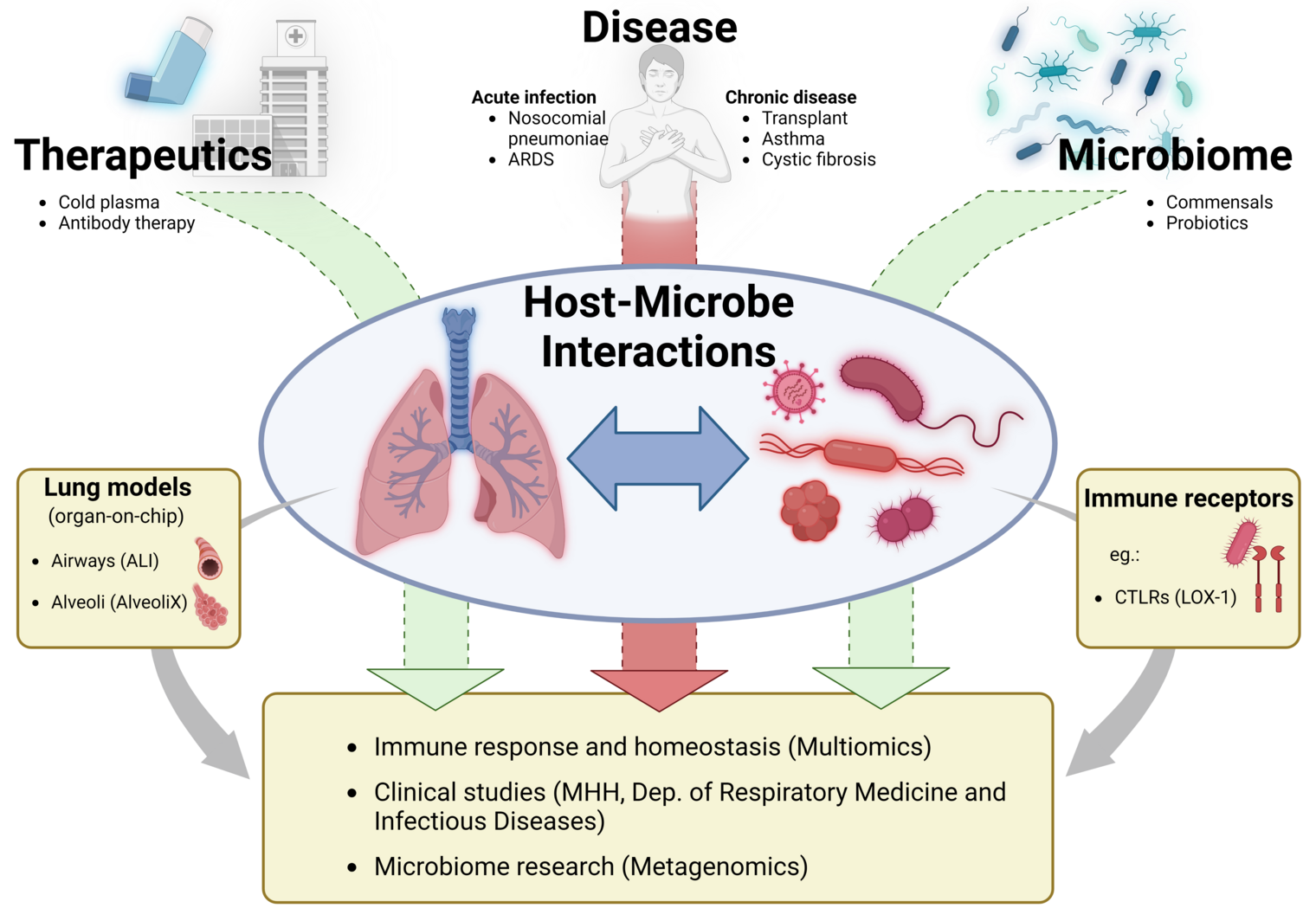
Die Forschungsgruppe „Dynamik respiratorischer Infektionen“ (DINF) beschäftigt sich mit der Untersuchung von Pathogen-Host-Interaktionen im Respirationstrakt, mit einem besonderen Fokus auf bakteriellen Pathogenen wie Staphylococcus aureus und Streptococcus pneumoniae sowie auf fungalen Pathogenen der Lunge, einschließlich Aspergillus-Arten. Unsere Forschung untersucht rezeptorvermittelte Mechanismen, durch die diese Pathogene mit dem pulmonalen Epithel und alveolären Makrophagen interagieren. Darüber hinaus analysieren wir die Rolle nicht-pathogener Kolonisierer der Lunge, die durch Modulation der Immunantwort über Rezeptorsignalwege sowohl das Verhalten der Pathogene als auch die angeborene Immunität des Wirts beeinflussen.
Unsere Arbeit stützt sich auf verschiedene methodische Ansätze, darunter Proteomics, Transcriptomics, mikrobiologische Analysen und Molekularbiologie. Air-Liquid-Interface (ALI)-Kulturen primärer Lungenepithelzellen stellen ein zentrales Modell dar, um zelluläre und molekulare Interaktionen zwischen Wirt und Pathogen zu untersuchen. Ergänzend verwenden wir Precision-Cut-Lung-Slices (PCLS) als ex vivo-Modell, das die Gewebearchitektur erhält und eine physiologisch relevante Untersuchung von Pathogendynamik und Wirtsantwort ermöglicht. Mittels 16S-rRNA- und Metagenom-Sequenzierung analysieren wir die Zusammensetzung und Funktion des Lungenmikrobioms unter gesunden und pathologischen Bedingungen.
Unser Ziel ist es, die molekularen Mechanismen zu identifizieren, die pulmonale Infektionen antreiben, und innovative therapeutische Strategien zu entwickeln, um das Lungenmikrobiom und die Immunantwort gezielt zu modulieren.
Darüber hinaus sollen neue Ansätze für innovative Strategien zur verbesserten Prävention und Behandlung von respiratorischen Infektionen entwickelt werden.
Unsere Forschung
Die Forschungsgruppe „Dynamik respiratorischer Infektionen“ (DINF) beschäftigt sich mit der Untersuchung von Pathogen-Host-Interaktionen im Respirationstrakt, mit einem besonderen Fokus auf bakteriellen Pathogenen wie Staphylococcus aureus und Streptococcus pneumoniae sowie auf fungalen Pathogenen der Lunge, einschließlich Aspergillus-Arten. Unsere Forschung untersucht rezeptorvermittelte Mechanismen, durch die diese Pathogene mit dem pulmonalen Epithel und alveolären Makrophagen interagieren. Darüber hinaus analysieren wir die Rolle nicht-pathogener Kolonisierer der Lunge, die durch Modulation der Immunantwort über Rezeptorsignalwege sowohl das Verhalten der Pathogene als auch die angeborene Immunität des Wirts beeinflussen.
Unsere Arbeit stützt sich auf verschiedene methodische Ansätze, darunter Proteomics, Transcriptomics, mikrobiologische Analysen und Molekularbiologie. Air-Liquid-Interface (ALI)-Kulturen primärer Lungenepithelzellen stellen ein zentrales Modell dar, um zelluläre und molekulare Interaktionen zwischen Wirt und Pathogen zu untersuchen. Ergänzend verwenden wir Precision-Cut-Lung-Slices (PCLS) als ex vivo-Modell, das die Gewebearchitektur erhält und eine physiologisch relevante Untersuchung von Pathogendynamik und Wirtsantwort ermöglicht. Mittels 16S-rRNA- und Metagenom-Sequenzierung analysieren wir die Zusammensetzung und Funktion des Lungenmikrobioms unter gesunden und pathologischen Bedingungen.
Unser Ziel ist es, die molekularen Mechanismen zu identifizieren, die pulmonale Infektionen antreiben, und innovative therapeutische Strategien zu entwickeln, um das Lungenmikrobiom und die Immunantwort gezielt zu modulieren.
Darüber hinaus sollen neue Ansätze für innovative Strategien zur verbesserten Prävention und Behandlung von respiratorischen Infektionen entwickelt werden.
Prof. Dr. med. Hortense Slevogt
Ein besseres Verständnis der Wirt-Mikroben-Interaktionen in der Lunge könnte uns helfen, neue therapeutische Strategien für eine Vielzahl von Atemwegsinfektionen zu entwickeln.

Prof. Dr. med. Hortense Slevogt hat sich als renommierte Forscherin und Ärztin auf dem Gebiet der Atemwegsinfektionen und Immunologie etabliert. Ihr akademischer Weg begann mit einem Studium der Medizin an der Freien Universität Berlin, während dessen sie ein besonderes Interesse an Infektionskrankheiten und deren Auswirkungen auf die Gesundheit der Atemwege entwickelte. Angetrieben von ihrer Leidenschaft, die komplizierten Abläufe des menschlichen Immunsystems zu verstehen, spezialisierte sie sich weiter auf Innere Medizin und Pulmologie und wurde zu einer anerkannten Expertin auf diesem Gebiet.
Im Jahr 2009 wurden Prof. Slevogts Leistungen durch den Abschluss ihrer Habilitation in Innerer Medizin an der Charité - Universitätsmedizin Berlin gewürdigt. Dies war ein bedeutender Meilenstein, der ihr Fachwissen und ihr tiefes Verständnis für die komplexe Natur schwerer Infektionen unterstrich. Einige Jahre später, im Jahr 2011, übernahm Prof. Slevogt die Leitung der Host Septomics Group am Universitätsklinikum Jena.
Im Jahr 2022 erhielt sie die W3-Professur an der Medizinischen Hochschule Hannover (MHH) und wurde Leiterin der Forschungsgruppe "Dynamik respiratorischer Infektionen" (DINF) am Helmholtz-Zentrum für Infektionsforschung (HZI) in Braunschweig. In dieser Funktion leitet sie translationale Projekte, bei denen sie mit renommierten akademischen und medizinischen Partnern der MHH zusammenarbeitet. Im Rahmen dieser Projekte untersucht Dr. Slevogt das Wirt-Erreger-Interaktom und klärt die Mechanismen der Immunantwort und deren Kontrolle auf.
Parallel zu ihrer Forschungstätigkeit widmet sich Dr. Slevogt der klinischen Praxis, um sicherzustellen, dass ihre Erkenntnisse direkt den Patienten zugutekommen. Als leitende Oberärztin für klinische Infektiologie in der Abteilung für Atemwegsmedizin und Infektionskrankheiten (MHH) trägt sie mit ihrer Expertise zur Diagnose und Behandlung von Atemwegsinfektionen bei.










Team

Ausgewählte Publikationen
Klassert TE #, Goyal S, Stock M, Driesch D, Hussain A, Berrocal-Almanza LC, Myakala R, Sumanlatha G, Valluri V, Ahmed N, Schumann RR, Flores C, Slevogt H §. (2018). AmpliSeq Screening of Genes Encoding the C-Type Lectin Receptors and Their Signaling Components Reveals a Common Variant in MASP1 Associated with Pulmonary Tuberculosis in an Indian Population. Front Immunol. 9:242. 10.3389/fimmu.2018.00242.
Lehmann R, Müller MM, Klassert TE, Driesch D, Stock M, Heinrich A, Conrad T, Moore C, Schier UK, Guthke R, Slevogt H §. (2018). Differential regulation of the transcriptomic and secretomic landscape of sensor and effector functions of human airway epithelial cells. Mucosal Immunol. 11(3):627-642. 10.1038/mi.2017.100.
Klassert TE #, Leistner R, Zubiria-Barrera C, Stock M, López M, Neubert R, Driesch D, Gastmeier P, Slevogt H §. (2021). Bacterial colonization dynamics and antibiotic resistance gene dissemination in the hospital environment after first patient occupancy: a longitudinal metagenetic study. Microbiome. 9(1):169. 10.1186/s40168-021-01109-7.
Klassert TE #, Zubiria-Barrera C, Neubert R, Stock M, Schneegans A, López M, Driesch D, Zakonsky G, Gastmeier P, Slevogt H §, Leistner R. (2022). Comparative analysis of surface sanitization protocols on the bacterial community structures in the hospital environment. Clin Microbiol Infect. 28(8):1105-1112. 10.1016/j.cmi.2022.02.032.
Müller MM, Baldauf C, Hornischer S, Klassert TE, Schneegans A, Behnert A, Pletz MW, Hagel S, Slevogt H §. (2023). Staphylococcus aureus induces tolerance in human monocytes accompanied with expression changes of cell surface markers. Front Immunol. 14:1046374. 10.3389/fimmu.2023.1046374.
Projekte
- Der Einfluss mikrobieller Kommensalen-Pathogen-Interaktionen auf die Gestaltung des immunologischen Milieus des menschlichen Respirationstrakts (RESIST)
- PlasmaCare: Analyse der Sicherheit der Anwendung von kaltem Plasma auf das menschliche Lungenepithel anhand komplexer Zellkulturmodelle
- Das Zusammenspiel von Candida albicans- und Aspergillus fumigatus-induzierten Signalen bei der rezeptorvermittelten Modulation der antimykotischen Immunantwort
- Entwicklung eines neuartigen Testkits für die Diagnostik einer Pneumozystis jirovecii-Infektion