Unsere Forschung
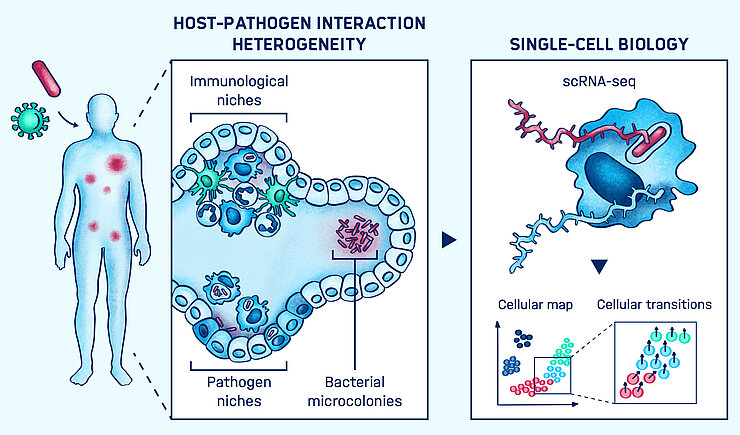
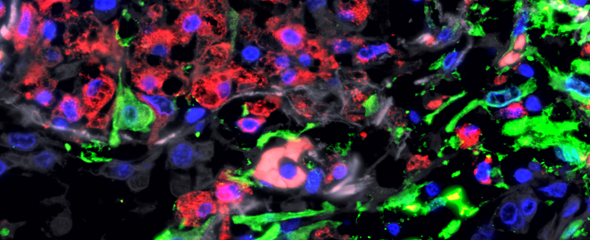
Wiederkehrende bakterielle Infektionen können von einer kleinen Subpopulation von Krankheitserregern verursacht werden, die in verschiedenen Zell- und Gewebetypen asymptomatisch überdauern. Diese als „Persister“ bezeichneten Bakterien sind aufgrund einer speziellen Zellorganisation vor dem Immunsystem des Wirtes geschützt und zeigen sich unempfindlich gegenüber Antibiotikagabe.
Die zellulären Mechanismen und bevorzugten Nischen der Pathogene sind weitestgehend unverstanden und scheinen äußerst vielfältig zu sein. So kann beispielsweise Salmonella spp in einer Vielzahl von Zelltypen, wie Makrophagen, neutrophilen Granulozyten, dendritischen Zellen und Epithelzellen, überdauern. Dass sich diese großen Zelltypen in zahlreiche Unterklassen einteilen lassen, hat erst in den letzten Jahren an Bedeutung gewonnen. Auch innerhalb eines infizierten Gewebes können sich viele Zellen vom Entzündungsherd lösen und in andere Gewebe streuen. In vivo Einzelzellstudien stellen daher eine essentielle Möglichkeit dar, die Heterogenität infizierter Zellen, deren Mikroumgebung und ihre Funktion analysieren zu können.
In unserer Arbeitsgruppe entwickeln und kombinieren wir Einzelzell-Transkriptom-Ananlysen in vivo und in vitro. Auf diesem Wege untersuchen wir die zelluläre Organisation innerhalb von Infektionsherden und betrachten funktionelle Auswirkungen auf den Krankheitsverlauf.
Die jüngste Entwicklung der genomweiten Trankriptom-Analyse auf Einzelzellebene bietet die Möglichkeit, parallel die Identität und Funktion von Zellen zu bestimmen. Darauf aufbauend haben wir Methoden der Einzelzell-RNA-Sequenzierung entwickelt, mit deren Hilfe wir Heterogenität in Immunprozessen analysieren können. So konnten wir beispielsweise bei Makrophagen aus dem Knochenmark von Mäusen, die mit Salmonella infiziert wurden, eine Subpopulation von Zellen beschreiben, die sich der Aktivierung der Entzündungsreaktion und Immunantwort entziehen konnte. Im nächsten Schritt möchten wir die Techniken der in vitro-Analyse in Zellkulturen auf Einzelzellanalysen in infizierten Geweben übertragen.
Unsere Forschung
Wiederkehrende bakterielle Infektionen können von einer kleinen Subpopulation von Krankheitserregern verursacht werden, die in verschiedenen Zell- und Gewebetypen asymptomatisch überdauern. Diese als „Persister“ bezeichneten Bakterien sind aufgrund einer speziellen Zellorganisation vor dem Immunsystem des Wirtes geschützt und zeigen sich unempfindlich gegenüber Antibiotikagabe.
Die zellulären Mechanismen und bevorzugten Nischen der Pathogene sind weitestgehend unverstanden und scheinen äußerst vielfältig zu sein. So kann beispielsweise Salmonella spp in einer Vielzahl von Zelltypen, wie Makrophagen, neutrophilen Granulozyten, dendritischen Zellen und Epithelzellen, überdauern. Dass sich diese großen Zelltypen in zahlreiche Unterklassen einteilen lassen, hat erst in den letzten Jahren an Bedeutung gewonnen. Auch innerhalb eines infizierten Gewebes können sich viele Zellen vom Entzündungsherd lösen und in andere Gewebe streuen. In vivo Einzelzellstudien stellen daher eine essentielle Möglichkeit dar, die Heterogenität infizierter Zellen, deren Mikroumgebung und ihre Funktion analysieren zu können.
In unserer Arbeitsgruppe entwickeln und kombinieren wir Einzelzell-Transkriptom-Ananlysen in vivo und in vitro. Auf diesem Wege untersuchen wir die zelluläre Organisation innerhalb von Infektionsherden und betrachten funktionelle Auswirkungen auf den Krankheitsverlauf.
Die jüngste Entwicklung der genomweiten Trankriptom-Analyse auf Einzelzellebene bietet die Möglichkeit, parallel die Identität und Funktion von Zellen zu bestimmen. Darauf aufbauend haben wir Methoden der Einzelzell-RNA-Sequenzierung entwickelt, mit deren Hilfe wir Heterogenität in Immunprozessen analysieren können. So konnten wir beispielsweise bei Makrophagen aus dem Knochenmark von Mäusen, die mit Salmonella infiziert wurden, eine Subpopulation von Zellen beschreiben, die sich der Aktivierung der Entzündungsreaktion und Immunantwort entziehen konnte. Im nächsten Schritt möchten wir die Techniken der in vitro-Analyse in Zellkulturen auf Einzelzellanalysen in infizierten Geweben übertragen.
Dr. Antoine-Emmanuel Saliba
Die Entwicklung der Einzelzell-Sequenzierung ist ein fantastischer wissenschaftlicher Fortschritt, vergleichbar mit der Genom-Sequenzierung. In absehbarer Zeit werden wir nicht nur in der Infektionsbiologie quantitative Voraussagen tätigen können, die auf Einzelzellanalysen beruhen.

Antoine-Emmanuel Saliba studierte Biochemietechnik an der INSA Toulouse (Frankreich) und promovierte in der Arbeitsgruppe von Jean-Louis Viovy am Institut Curie (Paris, Frankreich). Während dieser Zeit entwickelte er auf Mikrofluiden basierende Systeme zur Sortierung und Analyse seltener Subpopulationen von Krebszellen. Anschließend war er als EIPOD-Stipendiat am European Molecular Biology Laboratory (Heidelberg, Germany) tätig und an der Entwicklung innovativer systembiologischer Verfahren zur Analyse von Protein-Lipid-Interaktionen beteiligt. Danach schloss er sich als Senior Postdoc dem Labor von Jörg Vogel in Würzburg an und etablierte die Einzelzell-RNA-Sequenzierung zur Verfolgung des Schicksals individueller Zellen währen einer Infektion mit Salmonella enterica. Seit 2017 leitet er die Einzelzellanalyse-Arbeitsgruppe am HIRI.
Ausgewählte Publikationen
Cochain C*, Vafadarnejad E*, Arampatzi P, Pelisek J, Winkels H, Ley K, Wolf D, Saliba AE*, Zernecke A*
Single-cell RNA-seq reveals the transcriptional landscape and heterogeneity of aortic macrophages in murine atherosclerosis
Circulation Res 2018, DOI: 10.1161/CIRCRESAHA.117.312509
Saliba AE, C Santos S, Vogel J
New RNA-seq approaches for the study of bacterial pathogens
Curr Opin Microbiol 2017, 35:78-87
Saliba AE, Li L, Westermann AJ, Appenzeller S, Stapels DA, Schulte LN, Helaine S, Vogel J
Single-cell RNA-seq ties macrophage polarization to growth rate of intracellular Salmonella
Nature Microbiol 2016, 2:16206
Saliba AE, Vonkova I, Deghou S, Ceschia S, Tischer C, Kugler KG, Bork P, Ellenberg J, Gavin AC
A protocol for the systematic and quantitative measurement of protein-lipid interactions using the liposome-microarray-based assay
Nature Protoc 2016, 11(6): 1021-1038
Vonkova I*, Saliba AE*, Deghou S, Anand K, Ceschia S, Doerks T, Galih A, Kugler KG, Maeda K, Rybin V, van Noort V, Ellenberg J, Bork P, Gavin AC
Lipid Cooperativity as a General Membrane-Recruitment Principle for PH Domains
Cell Rep 2015, 12(9): 1519-1530