Genome Mining für Sekundärstoffe

Unsere Forschung
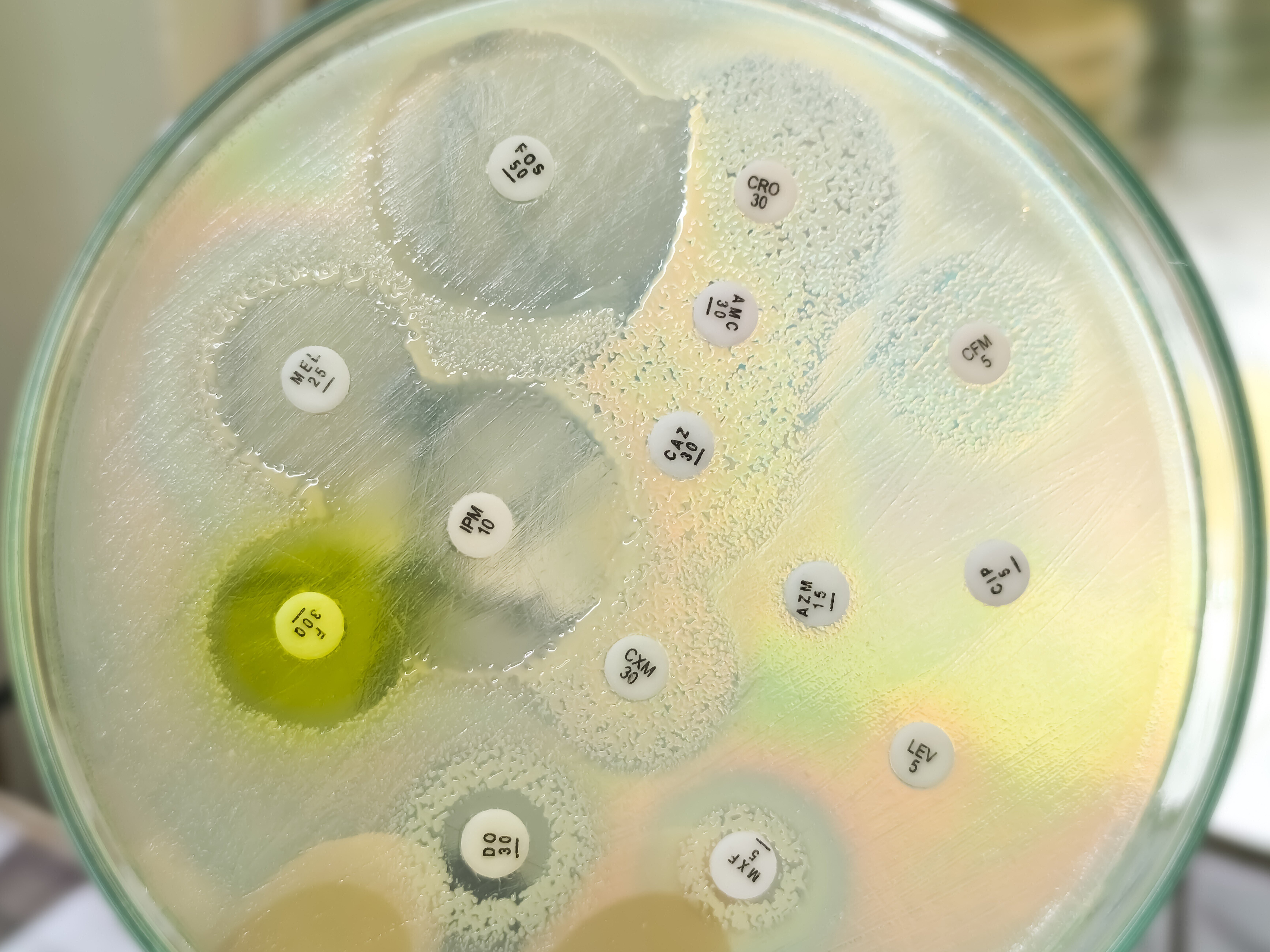
Die zunehmende Ausbreitung antimikrobieller Resistenzen stellt laut der Weltgesundheitsorganisation WHO eine der größten Bedrohungen für die globale Gesundheit, Lebensmittelsicherheit, sowie die wirtschaftliche Entwicklung dar. Während die Entwicklung von Resistenzen hauptsächlich durch den unsachgemäßen Gebrauch von Antibiotika verursacht wird, wird die Lage durch die sehr geringe Anzahl an neu entwickelten Antibiotika weiter verschärft. Daher ist die Nachfrage nach neuen, resistenzbrechenden Wirkstoffen höher denn je.
Naturstoffe sind eine wertvolle Quelle für die Entwicklung niedermolekularer Wirkstoffe, welche unter anderem bei Infektions- oder Krebserkrankungen eingesetzt werden können. Bereits heute handelt es sich bei einem großen Prozentsatz der auf den Markt gebrachten Wirkstoffe um Naturstoffe oder deren Derivate. Obwohl Mikroorganismen in der Vergangenheit bereits häufig als Naturstoffproduzenten in der Wirkstoffentwicklung verwendet wurden, zeigen moderne Genomanalysen, dass ihr Potential zur Produktion neuartiger Substanzen noch lange nicht erschöpft ist.
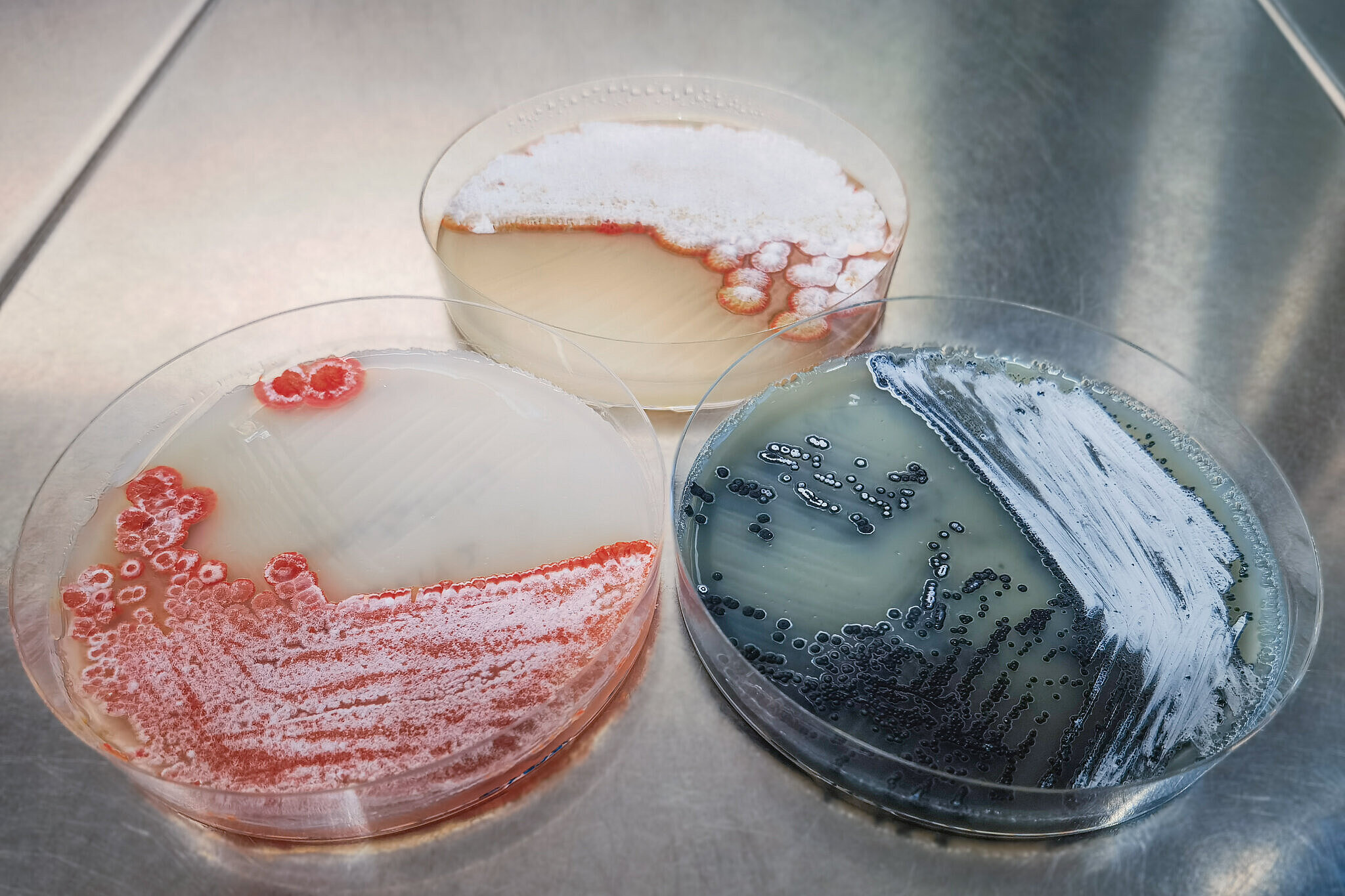
Eine der größten Herausforderungen bei der naturstoffbasierten Wirkstoffentwicklung ist die hohe Wiederentdeckungsrate. Die Entwicklung von Methoden zur gezielten Identifizierung neuer, bioaktiver Substanzen trägt daher einen großen Beitrag zur Bekämpfung antimikrobieller Resistenzen dar. Mit der zunehmenden Verfügbarkeit vollständiger Genomsequenzen von Naturstoffproduzenten zu Beginn des neuen Jahrtausends wurde klar, dass deren Potential zur Produktion strukturell unterschiedlicher Sekundärstoffe weitaus höher ist als bislang vermutet. Unter gewöhnlichen Laborbedingungen wird von Bakterien oftmals nur eine geringe Zahl an Naturstoffen hergestellt. Ein spannender Ansatz ist daher die Entwicklung von Strategien zur Aktivierung inaktiver Biosynthesewege mit unbekannter Funktion. Die Tatsache, dass dieser Ansatz in der Vergangenheit nur in wenigen Fällen zum Erfolg geführt hat zeigt, dass neue und effektivere Methoden dringend benötigt werden.
Unsere Arbeitsgruppe entwickelt Ideen und Methoden zur Entdeckung neuer Wirkstoffe aus Mikroorganismen. Unser Ziel ist es Naturstoffe zu entdecken, welche einen Beitrag zur Bekämpfung antimikrobieller Resistenzen leisten können. Wir verwenden Ansätze aus dem Bereich des genome mining um mikrobielle Wirkstoffe mit neuartigen und damit möglicherweise resistenzbrechenden Wirkmechanismen zu identifizieren. Weiterhin kommen Methoden wie direct cloning, genome editing oder biosensor technology zum Einsatz um das umfassende biosynthetische Potential bakterieller Genome zugänglich zu machen.
Unsere Forschung
Die zunehmende Ausbreitung antimikrobieller Resistenzen stellt laut der Weltgesundheitsorganisation WHO eine der größten Bedrohungen für die globale Gesundheit, Lebensmittelsicherheit, sowie die wirtschaftliche Entwicklung dar. Während die Entwicklung von Resistenzen hauptsächlich durch den unsachgemäßen Gebrauch von Antibiotika verursacht wird, wird die Lage durch die sehr geringe Anzahl an neu entwickelten Antibiotika weiter verschärft. Daher ist die Nachfrage nach neuen, resistenzbrechenden Wirkstoffen höher denn je.
Naturstoffe sind eine wertvolle Quelle für die Entwicklung niedermolekularer Wirkstoffe, welche unter anderem bei Infektions- oder Krebserkrankungen eingesetzt werden können. Bereits heute handelt es sich bei einem großen Prozentsatz der auf den Markt gebrachten Wirkstoffe um Naturstoffe oder deren Derivate. Obwohl Mikroorganismen in der Vergangenheit bereits häufig als Naturstoffproduzenten in der Wirkstoffentwicklung verwendet wurden, zeigen moderne Genomanalysen, dass ihr Potential zur Produktion neuartiger Substanzen noch lange nicht erschöpft ist.
Eine der größten Herausforderungen bei der naturstoffbasierten Wirkstoffentwicklung ist die hohe Wiederentdeckungsrate. Die Entwicklung von Methoden zur gezielten Identifizierung neuer, bioaktiver Substanzen trägt daher einen großen Beitrag zur Bekämpfung antimikrobieller Resistenzen dar. Mit der zunehmenden Verfügbarkeit vollständiger Genomsequenzen von Naturstoffproduzenten zu Beginn des neuen Jahrtausends wurde klar, dass deren Potential zur Produktion strukturell unterschiedlicher Sekundärstoffe weitaus höher ist als bislang vermutet. Unter gewöhnlichen Laborbedingungen wird von Bakterien oftmals nur eine geringe Zahl an Naturstoffen hergestellt. Ein spannender Ansatz ist daher die Entwicklung von Strategien zur Aktivierung inaktiver Biosynthesewege mit unbekannter Funktion. Die Tatsache, dass dieser Ansatz in der Vergangenheit nur in wenigen Fällen zum Erfolg geführt hat zeigt, dass neue und effektivere Methoden dringend benötigt werden.
Unsere Arbeitsgruppe entwickelt Ideen und Methoden zur Entdeckung neuer Wirkstoffe aus Mikroorganismen. Unser Ziel ist es Naturstoffe zu entdecken, welche einen Beitrag zur Bekämpfung antimikrobieller Resistenzen leisten können. Wir verwenden Ansätze aus dem Bereich des genome mining um mikrobielle Wirkstoffe mit neuartigen und damit möglicherweise resistenzbrechenden Wirkmechanismen zu identifizieren. Weiterhin kommen Methoden wie direct cloning, genome editing oder biosensor technology zum Einsatz um das umfassende biosynthetische Potential bakterieller Genome zugänglich zu machen.
Dr. Chengzhang Fu
Die Natur bietet vielfältige Möglichkeiten zur Herstellung neuartiger Wirkstoffe. Die Entwicklung von genome mining Ansätzen wird uns dabei helfen, das Potential kryptischer Biosynthesewege aus Bakterien für die Produktion neuartiger Sekundärstoffe zu nutzen.

Chengzhang Fu studierte Biotechnologie an der Universität von Zhejiang in China von 2002 bis 2006. 2011 promovierte er im Fachbereich Biochemie und Molekularbiologie am mikrobiologischen Institut der Chinesischen Akademie der Wissenschaften. Er kam 2012 als Alexander von Humboldt Fellow in die Gruppe von Rolf Müller an der Universität des Saarlandes. Diese Zusammenarbeit wurde ab 2014 am Helmholtz-Institut für Pharmazeutische Forschung Saarland (HIPS) fortgeführt. Seit 2019 ist Chengzhang Fu Nachwuchsgruppenleiter am HIPS im Rahmen des „Helmholtz International Labs“ Programmes. Seine Gruppe arbeitet an der Identifizierung und Verbesserung bislang unbekannter Biosynthesewege aus Bakterien und Umwelt-DNA.
Ausgewählte Publikationen
- Fu, C.; Auerbach, D.; Li, Y.; Scheid, U.; Luxenburger, E.; Garcia, R.; Irschik, H.; Müller, R. Solving the Puzzle of One-Carbon Loss in Ripostatin Biosynthesis. Angewandte Chemie (International ed. in English) 2017, 56 (8), 2192–2197. DOI: 10.1002/anie.201609950.
- Fu, C.; Keller, L.; Bauer, A.; Brönstrup, M.; Froidbise, A.; Hammann, P.; Herrmann, J.; Mondesert, G.; Kurz, M.; Schiell, M.; Schummer, D.; Toti, L.; Wink, J.; Müller, R. Biosynthetic Studies of Telomycin Reveal New Lipopeptides with Enhanced Activity. J. Am. Chem. Soc. 2015, 137 (24), 7692–7705. DOI: 10.1021/jacs.5b01794.
- Fu, C.; Sikandar, A.; Donner, J.; Zaburannyi, N.; Herrmann, J.; Reck, M.; Wagner-Döbler, I.; Koehnke, J.; Müller, R. The natural product carolacton inhibits folate-dependent C1 metabolism by targeting FolD/MTHFD. Nature communications 2017, 8 (1), 1529. DOI: 10.1038/s41467-017-01671-5
- Wang, Q.; Xie, F.; Tong, Y.; Habisch, R.; Yang, B.; Zhang, L.; Müller, R.; Fu, C. Dual-function chromogenic screening-based CRISPR/Cas9 genome editing system for actinomycetes. Applied microbiology and biotechnology 2020, 104 (1), 225–239. DOI: 10.1007/s00253-019-10223-4.
- Wang, T.; Wang, X.; Zhao, H.; Huo, L.; Fu, C. Uncovering a Subtype of Microviridins via the Biosynthesis Study of FR901451. ACS chemical biology 2022, 17 (12), 3489–3498. DOI: 10.1021/acschembio.2c00688.
Eine komplette Liste aller Publikationen finden Sie auf der HIPS-Webseite.