Integrative Informatik der Infektionsbiologie

Unsere Forschung
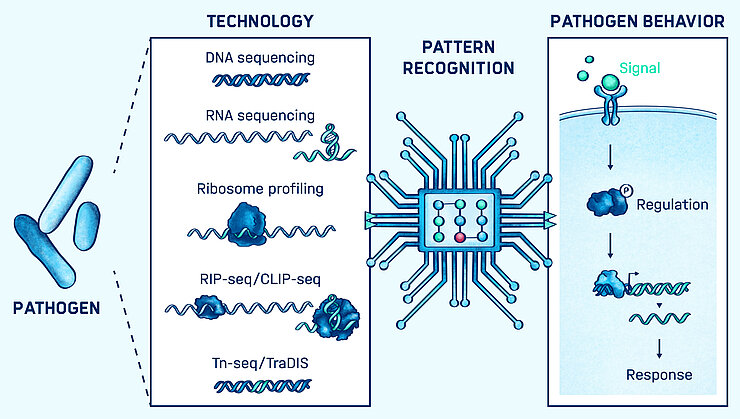
Das Verhalten einzelner Zellen während komplexer Infektionsprozesse kann auf Genom-Ebene durch funktionelle Hochdurchsatz-Technologien verfolgt werden. Dadurch lassen sich beispielsweise mechanistische Interaktionen zwischen Bakterien und ihren Wirtszellen analysieren.
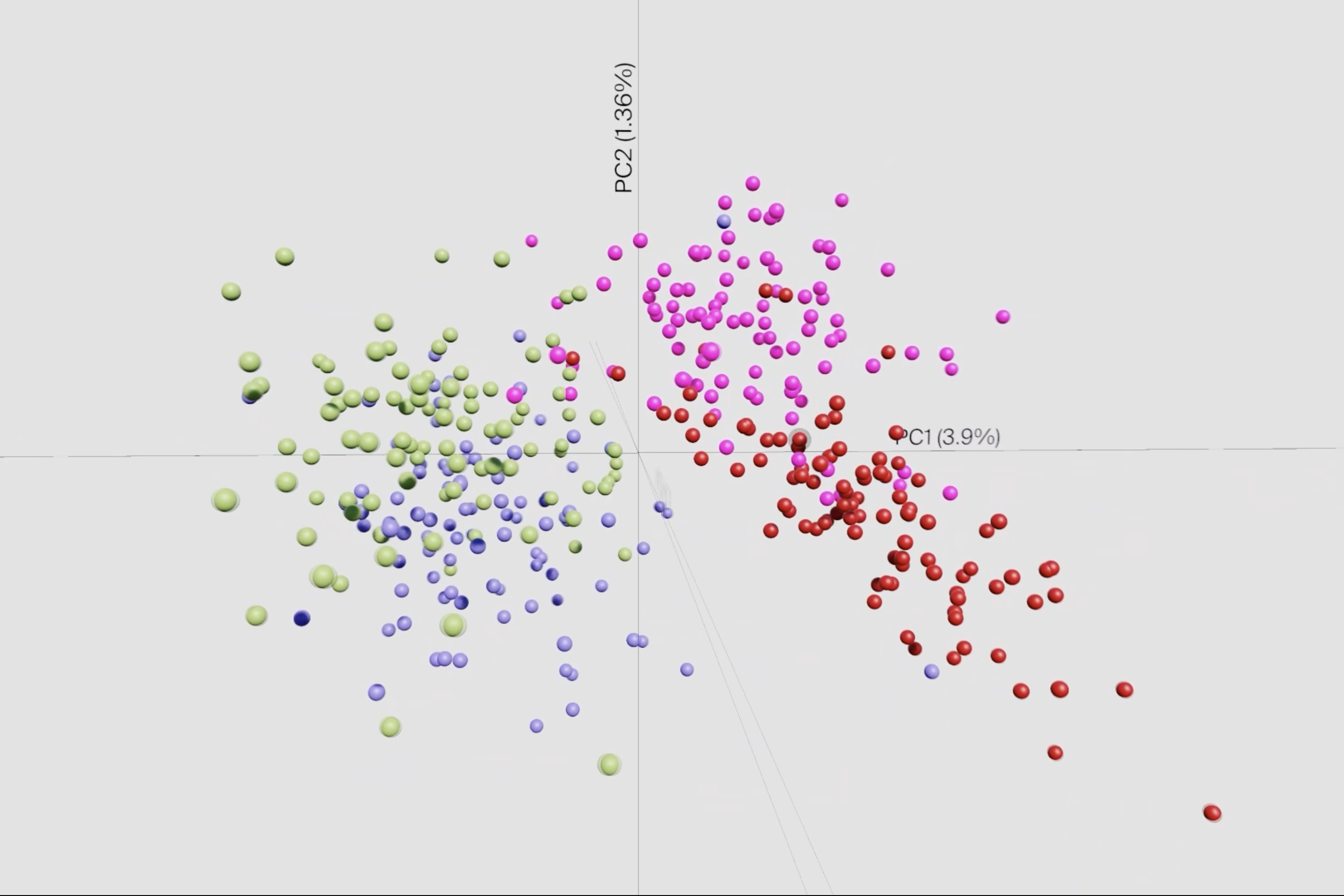
Die Herausforderung stellt jedoch die Integration und standardisierte Interpretation der vielschichtigen Datensätze dar. Um aus den genomischen und post-genomischen Daten biologische Erkenntnisse gewinnen zu können, nutzen wir modernste Data Science Technologien (Maschinelles Lernen, Visualisierung und statistische Modellierung). Unser spezieller Fokus liegt dabei auf RNA-basierten Regulationen in Bakterien, sowie der Rolle nicht-kodierender RNAs bei Wirt-Pathogen-Interaktionen und während der Evolution von Pathogenen.
Unsere Forschung
Das Verhalten einzelner Zellen während komplexer Infektionsprozesse kann auf Genom-Ebene durch funktionelle Hochdurchsatz-Technologien verfolgt werden. Dadurch lassen sich beispielsweise mechanistische Interaktionen zwischen Bakterien und ihren Wirtszellen analysieren.
Die Herausforderung stellt jedoch die Integration und standardisierte Interpretation der vielschichtigen Datensätze dar. Um aus den genomischen und post-genomischen Daten biologische Erkenntnisse gewinnen zu können, nutzen wir modernste Data Science Technologien (Maschinelles Lernen, Visualisierung und statistische Modellierung). Unser spezieller Fokus liegt dabei auf RNA-basierten Regulationen in Bakterien, sowie der Rolle nicht-kodierender RNAs bei Wirt-Pathogen-Interaktionen und während der Evolution von Pathogenen.
Jun. Prof. Dr. Lars Barquist
Moderne Hochdurchsatz-Technologien haben die Art und Weise, in der biologische Forschung betrieben wird, massiv verändert und bedürfen neuer rechnergestützter und statistischer Analyse-Werkzeuge. Unser Ziel ist es, diese Technologien zu erschließen, um so neue Einsichten in die Pathogenese von Bakterien zu ermöglichen.

Lars Barquist studierte Biomathematik an der Rutgers University (New Jersey, USA) und arbeitete anschließend an der University of California (Berkeley, USA) im Bereich Biotechnik. Im Jahr 2014 erhielt er seinen Doktortitel von der University of Cambridge (UK) für seine Arbeit über vergleichende funktionelle Genomik von Pathogenen am unabhängigen Wellcome Trust Sanger Institute. Von 2014 bis 2016 erhielt er ein Forschungsstipendium der Alexander-von-Humboldt-Stiftung und arbeitete am Institut für Molekulare Infektionsbiologie in Würzburg.
Ausgewählte Publikationen
Barquist L, Vogel J
Accelerating discovery and functional analysis of small RNAs with new technologies
Annu Rev Genet 2015, 49: 367-394
Wheeler NE, Gardner PP, Barquist L
Machine learning identifies signatures of host adaptation in the bacterial pathogen Salmonella enterica
PLOS Genetics 2018
Barquist L, Mayho M, Cummins C, Cain AK, Boinett CJ, Page AJ, Langridge GC, Quail MA, Keane JA, Parkhill J
The TraDIS toolkit: sequencing and analysis for dense transposon mutant libraries
Bioinformatics 2016, 32(7): 1109-1111
Wheeler NE, Barquist L, Kingsley RA, Gardner PP
A profile-based method for identifying functional divergence of orthologous genes in bacterial genes
Bioinformatics 2016, 32(23): 3566-3574
Westermann AJ, Förstner KU, Amman F, Barquist L, Chao Y, Schulte LN, Müller L, Reinhardt R, Stadler PF, Vogel J
Dual RNA-seq unveils noncoding RNA functions in host-pathogen interactions
Nature 2016, 529(7587): 496-501