Komplexe in Phageninfizierten Zellen

Unsere Forschung
Phagen infizieren Bakterien, manipulieren ihre zelluläre Physiologie und lösen schließlich die Zellen auf. Viele Phagen werden durch Integration in das bakterielle Genom oder Koexistenz in der Population zu einem integralen Bestandteil von Bakterien, während andere eine ganze Bakterienpopulation auflösen können. Dies führt seit Milliarden von Jahren zu einem anhaltenden Konflikt zwischen Phagen und Bakterien. Die zyklischen Lyse- und Resistenz-Ereignisse zwischen Phagen und Bakterien haben die Evolution geprägt und unvorhersehbare Maschinen hervorgebracht, die z.B. für bahnbrechende Technologien wie CRISPR-Cas genutzt werden können. Die Phagen-Wirt-Schnittstelle ist der vielfältigste Sequenzraum im Protein-Universum und hier können Entdeckungen gemacht werden.
Unsere Gruppe interessiert sich besonders für Jumbo-Phagen, die Genomgrößen von >200 kB haben und Hunderte von Faktoren mit unbekannter Funktion kodieren. Diese Phagen replizieren sich schnell, bilden zelluläre Strukturen wie in eukaryotischen Zellen und verfügen über ein riesiges Repertoire an Funktionen, um die Kontrolle über ihren Wirt zu übernehmen. Dies ist ein Spezies-spezifischer Prozess, der Anpassung und Einfallsreichtum seitens des Phagen erfordert, um zelluläre Immunität oder Stress zu überwinden. Unser Ziel ist es, die Mechanismen der Phagen-Wirtsübernahme aufzuklären und ihre molekularen Mechanismen funktionell zu charakterisieren. Um Phagen-Faktoren zu identifizieren die mit Produktionsfabriken in Zellen interagieren, verwenden wir Multi-Omics-Ansätze und zur Aufklärung der molekularen und strukturellen Mechanismen hinter ihrer Rolle wenden wir Kryo-Elektronenmikroskopie (cryo-EM) und biochemische Methoden an. Unser Ziel ist es, diese Faktoren oder ihre Mechanismen in neuartige Therapien zur Behandlung von antibiotikaresistenten Krankheitserregern wie Pseudomonas zu übertragen.
Komplexe in phagen-infizierten Zellen
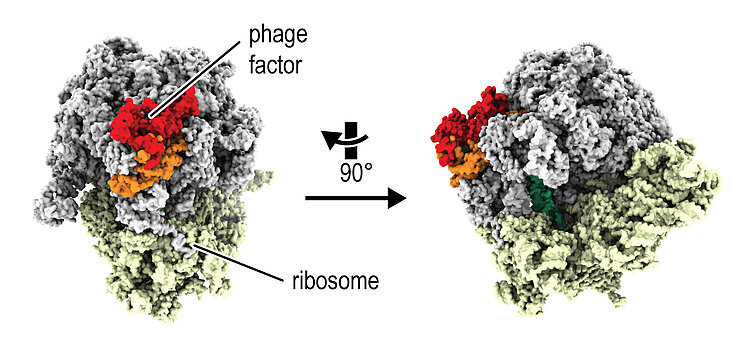
Wir verwenden Multi-Omics-Techniken, um Komplexe zwischen RNA, DNA und Proteinen in phageninfizierten Zellen zu analysieren. Wir haben kürzlich Phagen-Faktoren identifiziert, die direkt die Proteinproduktion adressieren, einen Mega-Komplex aus Proteinen und RNA, das Ribosom. Um die molekularen Mechanismen dieser Faktoren zu untersuchen, verwenden wir Strukturstudien wie die Einzelpartikel-cryo-EM und biochemische Ansätze. Unser Ziel ist es, zu verstehen, wie Phagen die Stressreaktion bei der Proteinproduktion modulieren.
Zelluläre Strukturen in phagen-infizierten Zellen
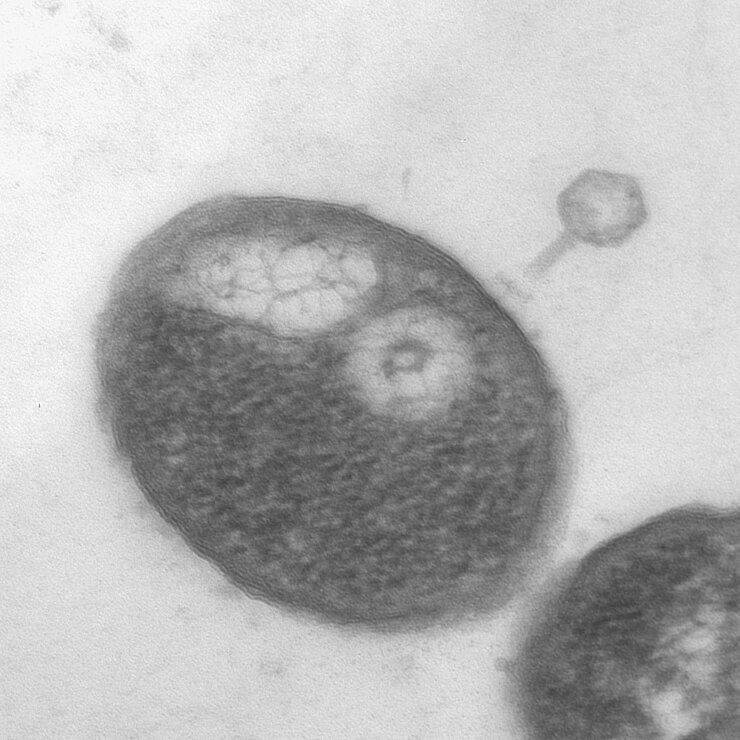
Beim ersten Kontakt injizieren Phagen ihr genetisches Material in den Wirt. Bemerkenswerterweise bilden einige Jumbo-Phagen der Familie Chimalliviridae selbstgemachte zelluläre Kompartimente, die ihr Genom schützen, aber auch als Produktionsstätten dienen. So wird beispielsweise ein protein-basierter Phagen-Kern gebildet, der zur Steuerung der Phagen-Produktion und Assemblierung verwendet wird. Wir sind neugierig darauf, zu untersuchen, wie sich Proteine und RNAs mit den von Phagen hergestellten zellulären Strukturen verbinden und dem Phagen-Replikationszyklus eine räumliche Komplexitätsebene hinzufügen. In unseren Studien freuen wir uns darauf, neue verborgene zelluläre Strukturen zu entdecken, die für künstliche Kompartimente in biotechnologischen und medizinischen Anwendungen genutzt werden könnten.
Unsere Forschung
Phagen infizieren Bakterien, manipulieren ihre zelluläre Physiologie und lösen schließlich die Zellen auf. Viele Phagen werden durch Integration in das bakterielle Genom oder Koexistenz in der Population zu einem integralen Bestandteil von Bakterien, während andere eine ganze Bakterienpopulation auflösen können. Dies führt seit Milliarden von Jahren zu einem anhaltenden Konflikt zwischen Phagen und Bakterien. Die zyklischen Lyse- und Resistenz-Ereignisse zwischen Phagen und Bakterien haben die Evolution geprägt und unvorhersehbare Maschinen hervorgebracht, die z.B. für bahnbrechende Technologien wie CRISPR-Cas genutzt werden können. Die Phagen-Wirt-Schnittstelle ist der vielfältigste Sequenzraum im Protein-Universum und hier können Entdeckungen gemacht werden.
Unsere Gruppe interessiert sich besonders für Jumbo-Phagen, die Genomgrößen von >200 kB haben und Hunderte von Faktoren mit unbekannter Funktion kodieren. Diese Phagen replizieren sich schnell, bilden zelluläre Strukturen wie in eukaryotischen Zellen und verfügen über ein riesiges Repertoire an Funktionen, um die Kontrolle über ihren Wirt zu übernehmen. Dies ist ein Spezies-spezifischer Prozess, der Anpassung und Einfallsreichtum seitens des Phagen erfordert, um zelluläre Immunität oder Stress zu überwinden. Unser Ziel ist es, die Mechanismen der Phagen-Wirtsübernahme aufzuklären und ihre molekularen Mechanismen funktionell zu charakterisieren. Um Phagen-Faktoren zu identifizieren die mit Produktionsfabriken in Zellen interagieren, verwenden wir Multi-Omics-Ansätze und zur Aufklärung der molekularen und strukturellen Mechanismen hinter ihrer Rolle wenden wir Kryo-Elektronenmikroskopie (cryo-EM) und biochemische Methoden an. Unser Ziel ist es, diese Faktoren oder ihre Mechanismen in neuartige Therapien zur Behandlung von antibiotikaresistenten Krankheitserregern wie Pseudomonas zu übertragen.
Komplexe in phagen-infizierten Zellen
Wir verwenden Multi-Omics-Techniken, um Komplexe zwischen RNA, DNA und Proteinen in phageninfizierten Zellen zu analysieren. Wir haben kürzlich Phagen-Faktoren identifiziert, die direkt die Proteinproduktion adressieren, einen Mega-Komplex aus Proteinen und RNA, das Ribosom. Um die molekularen Mechanismen dieser Faktoren zu untersuchen, verwenden wir Strukturstudien wie die Einzelpartikel-cryo-EM und biochemische Ansätze. Unser Ziel ist es, zu verstehen, wie Phagen die Stressreaktion bei der Proteinproduktion modulieren.
Zelluläre Strukturen in phagen-infizierten Zellen
Beim ersten Kontakt injizieren Phagen ihr genetisches Material in den Wirt. Bemerkenswerterweise bilden einige Jumbo-Phagen der Familie Chimalliviridae selbstgemachte zelluläre Kompartimente, die ihr Genom schützen, aber auch als Produktionsstätten dienen. So wird beispielsweise ein protein-basierter Phagen-Kern gebildet, der zur Steuerung der Phagen-Produktion und Assemblierung verwendet wird. Wir sind neugierig darauf, zu untersuchen, wie sich Proteine und RNAs mit den von Phagen hergestellten zellulären Strukturen verbinden und dem Phagen-Replikationszyklus eine räumliche Komplexitätsebene hinzufügen. In unseren Studien freuen wir uns darauf, neue verborgene zelluläre Strukturen zu entdecken, die für künstliche Kompartimente in biotechnologischen und medizinischen Anwendungen genutzt werden könnten.
Die Phagen-Wirt-Schnittstelle ist der vielfältigste Sequenzraum im Protein-Universum, der voller unvorhersehbarer Entdeckungen steckt.
Dr. Milan Gerovac
Dr. Milan Gerovac studierte Biochemie an der Goethe-Universität in Frankfurt am Main. In seiner Doktorarbeit bei Prof. Robert Tampé an der Goethe-Universität untersuchte er Kontrollmechanismen und Komplexe in der Proteinproduktion. Anschließend wechselte er als PostDoc an die Julius-Maximilians-Universität und das Helmholtz-Institut für RNA-basierte Infektionsforschung in Würzburg, wo er mit Jörg Vogel Methoden zur Entdeckung und Charakterisierung von RNA-bindenden Proteinen entwickelte und Erfahrungen in der Mikrobiologie mit verschiedenen Krankheitserregern sammelte. Er wechselte zur Erforschung der Phagen-Biologie, indem er RNA-Protein-Komplexe in mit phageninfizierten Zellen untersuchte, und entwickelte eine Faszination für den dunklen Protein-Raum in Phagen-Genomen, wobei er Phagen-Faktoren entdeckte, die auf die Ribosomen des Wirts abzielen. Im Jahr 2024 startete Gerovac die Nachwuchsgruppe Komplexe in phageninfizierten Zellen (CPIC) am HZI mit dem Ziel, Phagen-Mechanismen zu entdecken die genutzt werden können für neuartigen therapeutische Ansätze zur Behandlung von Infektionen.
Team
Wir sind immer auf der Suche nach neuen Mitgliedern für unser Team und freuen uns, wenn Sie uns kontaktieren und uns Ihre Bewerbung schicken.
Das Labor befindet sich derzeit im Aufbau und das Team wird zusammengestellt. Die Teammitglieder werden in Kürze bekannt gegeben.

Ausgewählte Publikationen
Gerovac M, Ðurica-Mitić S, Buhlmann L, Rech V, Zhu Y, Carien S, Popella L, Vogel J (2024)
Non-genetic messenger RNA silencing reveals essential genes in phage-host interplay.
bioRxiv, doi: 10.1101/2024.07.31.605949
Putzeys L & Wicke L & Brandão A, Boon M, Pires DP, Azeredo J, Vogel J, Lavigne R, Gerovac M (2024)
Exploring the transcriptional landscape of phage-host interactions using novel high-throughput approaches. Current Opinion in Microbiology 77, 102419. doi: 10.1016/j.mib.2023.102419
Gerovac M, Chihara K, Wicke L, Böttcher B, Lavigne R, Vogel J (2024)
Phage proteins target and co-opt host ribosomes immediately upon infection. Nature Microbiology 9, 787-800. doi: 10.1038/s41564-024-01616-x
Gerovac M, El Mouali Y, Kuper J, Kisker C, Barquist L, Vogel J (2020)
Global discovery of bacterial RNA-binding proteins by RNase-sensitive gradient profiles reports a new FinO domain protein. RNA 26, 1448-63. doi: 10.1261/rna.076992.120
*Heuer A & *Gerovac M, Schmidt C, Trowitzsch S, Preis A, Kötter P, Berninghausen O, Becker T, Beckmann R & Tampé R (2017) Structure of the 40S–ABCE1 post-splitting complex in ribosome recycling and translation initiation. Nature Structural & Molecular Biology 24, 453-60. doi: 10.1038/nsmb.3396
Technologieangebote
Folgende Technologien wurden von der Forschungsgruppe "Komplexe in Phageninfizierten Zellen" entwickelt und zum Patent angemeldet:
Novel ASO-Based mRNA Silencing Technology for Phage-Host Interaction Research
Newsroom
Sie haben Interesse an einer Bachelor-, Master-, Doktorarbeit? Wir freuen uns auf Ihre Anfrage!