LncRNA und Infektionsbiologie

Unsere Forschung
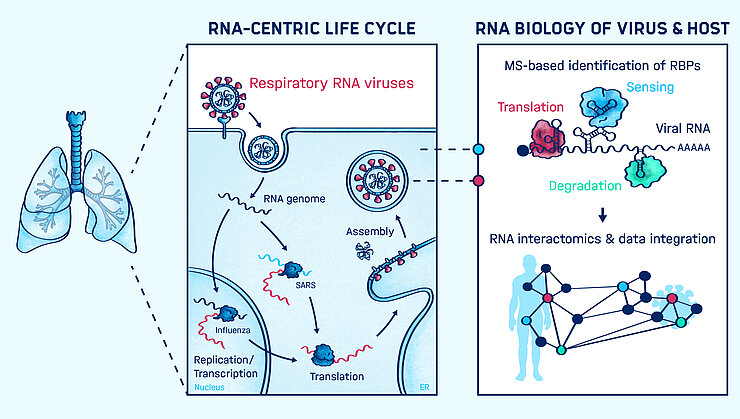
Für eine effektive Bekämpfung von Pathogenen muss die infizierte Wirtszelle dazu in der Lage sein ihre Genexpressionsprogramme dynamisch zu regulieren und eine geeignete Wirtsantwort zu steuern. Neben Boten-RNAs, die als Vorlage für die Synthese von Proteinen dienen, umfasst die zelluläre Wirtsantwort auch zahlreiche lange nicht-kodierenden RNAs (lncRNAs), die aktiv transkribiert und spezifisch reguliert werden. Da lncRNAs nicht für Proteine kodieren und stattdessen verschiedenste strukturelle oder regulatorische Funktionen haben können, ist die Entschlüsselung von physiologischen Funktionen und biochemischen Mechanismen einzelner lncRNA Regulatoren eine große Herausforderung. Folglich konnte die Rolle der meisten lncRNAs im Infektionsprozess bisher nicht gelöst werden.
Bahnbrechende Entwicklungen auf dem Feld der DNA Sequenziertechnologie haben es ermöglicht RNA Expressionsprogramme, die Teil der Wirtsantwort sind, systematisch zu charakterisieren. Die hieraus gewonnene Erkenntnis, dass Säugetiergenome hunderte bis tausende Pathogen-induzierter lncRNAs exprimieren, kann als molekulare Schatztruhe für die Entdeckung neuer genregulatorischer Mechanismen in der Wirtsantwort angesehen werden.
Das Ziel unserer Forschungsgruppe ist es den genetischen Code von lncRNA Regulatoren in Infektionskrankheiten zu entschlüsseln. Hierzu charakterisieren wir die Interaktionen von lncRNAs mit anderen Biomolekülen (RNA, DNA, Proteine) quantitativ und entschlüsseln assoziierte Sequenz- oder Strukturelemente. In diesem Zusammenhang, klären wir die Zusammensetzung von lncRNA-Protein Komplexen auf und bestimmen welche Interaktionen für die korrekte Funktion von lncRNA Regulatoren von Bedeutung sind.
Ein Schwerpunkt unserer Arbeit stellt hierbei die Erforschung von RNA-abhängigen Proteinkomplexen dar. Wie kürzlich von uns beschrieben, können lncRNAs direkt gebundene Proteine spezifisch regulieren, um unter anderem die Bildung von regulatorisch wichtigen Proteinkomplexen zu kontrollieren. Unsere Forschungsgruppe verwendet und entwickelt hierzu neuartige Technologien, die es erlauben direkte Interaktionen von einzelnen RNAs mit hoher Auflösung und in quantitativer Art und Weiße zu erfassen. Wir hoffen, dass die Erforschung von RNA-basierten Regulationsmechanismen dazu beitragen wird Infektionskrankheiten effektiver bekämpfen zu können.
Unsere Forschung
Für eine effektive Bekämpfung von Pathogenen muss die infizierte Wirtszelle dazu in der Lage sein ihre Genexpressionsprogramme dynamisch zu regulieren und eine geeignete Wirtsantwort zu steuern. Neben Boten-RNAs, die als Vorlage für die Synthese von Proteinen dienen, umfasst die zelluläre Wirtsantwort auch zahlreiche lange nicht-kodierenden RNAs (lncRNAs), die aktiv transkribiert und spezifisch reguliert werden. Da lncRNAs nicht für Proteine kodieren und stattdessen verschiedenste strukturelle oder regulatorische Funktionen haben können, ist die Entschlüsselung von physiologischen Funktionen und biochemischen Mechanismen einzelner lncRNA Regulatoren eine große Herausforderung. Folglich konnte die Rolle der meisten lncRNAs im Infektionsprozess bisher nicht gelöst werden.
Bahnbrechende Entwicklungen auf dem Feld der DNA Sequenziertechnologie haben es ermöglicht RNA Expressionsprogramme, die Teil der Wirtsantwort sind, systematisch zu charakterisieren. Die hieraus gewonnene Erkenntnis, dass Säugetiergenome hunderte bis tausende Pathogen-induzierter lncRNAs exprimieren, kann als molekulare Schatztruhe für die Entdeckung neuer genregulatorischer Mechanismen in der Wirtsantwort angesehen werden.
Das Ziel unserer Forschungsgruppe ist es den genetischen Code von lncRNA Regulatoren in Infektionskrankheiten zu entschlüsseln. Hierzu charakterisieren wir die Interaktionen von lncRNAs mit anderen Biomolekülen (RNA, DNA, Proteine) quantitativ und entschlüsseln assoziierte Sequenz- oder Strukturelemente. In diesem Zusammenhang, klären wir die Zusammensetzung von lncRNA-Protein Komplexen auf und bestimmen welche Interaktionen für die korrekte Funktion von lncRNA Regulatoren von Bedeutung sind.
Ein Schwerpunkt unserer Arbeit stellt hierbei die Erforschung von RNA-abhängigen Proteinkomplexen dar. Wie kürzlich von uns beschrieben, können lncRNAs direkt gebundene Proteine spezifisch regulieren, um unter anderem die Bildung von regulatorisch wichtigen Proteinkomplexen zu kontrollieren. Unsere Forschungsgruppe verwendet und entwickelt hierzu neuartige Technologien, die es erlauben direkte Interaktionen von einzelnen RNAs mit hoher Auflösung und in quantitativer Art und Weiße zu erfassen. Wir hoffen, dass die Erforschung von RNA-basierten Regulationsmechanismen dazu beitragen wird Infektionskrankheiten effektiver bekämpfen zu können.
Prof. Dr. Mathias Munschauer
Die Entschlüsselung von RNA-basierten Regulationsmechanismen wird es ermöglichen kodierende und nicht-kodierende RNAs für die bessere Behandlung von Infektionskrankheiten einzusetzen.

Mathias Munschauer hat ein naturwissenschaftliches Studium im Fach Biotechnologie an der Hochschule Mannheim absolviert. Schon vor Studienabschluss begann er im Labor von Thomas Tuschl an der Rockefeller University in New York (USA) an RNA zu forschen. Seine Abschlussarbeit zu dem RNA-bindenden Protein FMRP, absolvierte er unter Anleitung von Thomas Tuschl in New York. Seine Promotion führte er im Rahmen eines internationalen Doktoranden Programms sowohl am Max-Delbrück Center für Molekulare Medizin in Berlin (unter Anleitung von Markus Landthaler) als auch an der New York University in den USA (unter Anleitung von Christine Vogel) durch.
Im Labor von Markus Landthaler entwickelte Mathias Munschauer Schlüsseltechnologien, die es erstmalig erlaubten, alle RNA-bindenden Proteine in menschlichen Zellen zu identifizieren und gleichzeitig die von Proteinen kontaktierten RNA Sequenzregionen zu charakterisieren. Für sein Postdoktorat zog es Mathias Munschauer in das Labor von Eric Lander am Broad Institute of MIT and Harvard in den USA. Hier erforschte er die Funktionsmechanismen von langen nicht-kodierenden RNAs und identifizierte einen bis dahin unbekannten RNA-abhängigen Topoisomerase Komplex. Seit 2019 leitet er am Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) in Würzburg eine durch die Helmholtz-Gemeinschaft geförderte Nachwuchs-Forschungsgruppe.
Ausgewählte Publikationen
Munschauer, M.#, Nguyen, C.T., Sirokman, K., Hartigan C. R., Hogstrom, L., Engreitz, J. M, Fulco, C. P., Subramanian V., Chen, J., Ulirch J. C., Schenone, M. Guttman, M., Carr, S.A. Lander, E. S.# (2018). The NORAD lncRNA assembles a topoisomerase complex critical for genome stability. Nature, 561(7721):132-136. doi.org/10.1038/s41586-018-0453-z
Munschauer, M. and Vogel, J. (2018) Nuclear lncRNA stabilization in the host response to bacterial infection. EMBO J, 37(13). pii: e99875. doi.org/10.15252/embj.201899875.
Khajuria R.K., Munschauer, M., Ulirsch J.C., Fiorini C., Ludwig L.S., McFarland S.K., Abdulhay N.J., Specht H., Keshishian H., Mani D.R., Jovanovic M., Ellis S.R., Fulco C.P., Engreitz J.M., Schütz S., Lian J., Gripp K.W., Weinberg O.K., Pinkus G.S., Gehrke L., Regev A., Lander E.S., Gazda H.T., Lee W.Y., Panse V.G., Carr S.A., Sankaran V.G. (2018). Ribosome Levels Selectively Regulate Translation and Lineage Commitment in Human Hematopoiesis. Cell, 173(1):90-103.e19. doi.org/10.1016/j.cell.2018.02.036
Fulco, C. P., Munschauer, M., Anyoha, R., Munson, G., Grossman, S. R., Perez, E. M., Kane, M., Cleary B., Lander E. S., Engreitz E. M. (2016). Systematic mapping of functional enhancer-promoter connections with CRISPR interference. Science, 354(6313):769-773. doi.org/10.1126/science.aag2445
Baltz, A.G.*, Munschauer, M.*, Schwanhaeusser, B., Vasile, A., Murakawa, Y., Schueler, M., Youngs, N., Penfold-Brown, D., Drew, K., Milek, M., Wyler, E., Bonneau, R., Selbach, M., Dieterich, C., and Landthaler, M. (2012). The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Molecular Cell 46(5):674-90. doi.org/10.1016/j.molcel.2012.05.021