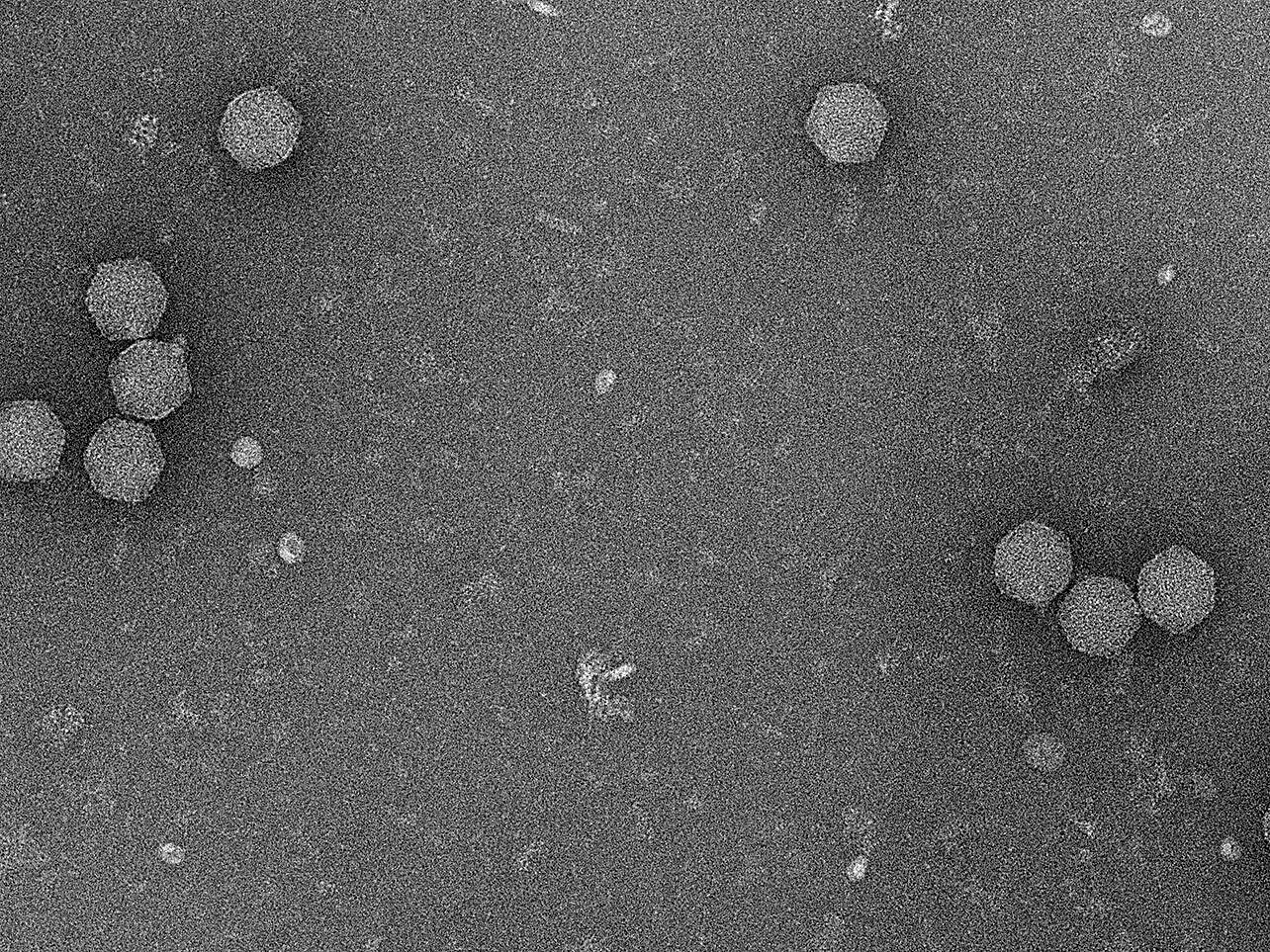
Molekulare Grundlagen von RNA-Phagen

Unsere Forschung
Angesichts der zunehmenden Bedrohung durch antibiotikaresistente Bakterien sind neue Behandlungsmöglichkeiten für bakterielle Infektionen dringend erforderlich. Phagen als die natürlichen Gegenspieler von Bakterien könnten Abhilfe schaffen. Hierfür ist es jedoch entscheidend, geeignete Phagen zu identifizieren und ihre Funktionsweise zu entschlüsseln. RNA-Phagen sind – im Gegensatz zu DNA-Phagen – wenig erforscht. Ihre Charakterisierung wird dazu beitragen, das volle Potenzial von Phagen bei der Entwicklung neuartiger antimikrobieller Therapien auszuschöpfen und birgt die Chance, innovative molekulare Werkzeuge und Technologien zu konzipieren.
Die Arbeitsgruppe von Jens Hör untersucht die molekularen Grundlagen dieser RNA-Phagen im Kontext der Phagen-Wirt-Interaktionen. Ihre Forschung zielt darauf ab, zu ergründen, wie RNA-Phagen ihre Wirte während der Infektion unter ihre Kontrolle bringen und wie die Wirte sich gegen die eindringenden Phagen verteidigen. Außerdem isoliert das Hör-Labor neue RNA-Phagen und charakterisiert sie funktionell.
Die Gruppe nutzt sowohl klassische biochemische und molekularbiologische Methoden als auch modernste Hochdurchsatzverfahren, um die besondere Lebensweise von RNA-Phagen im Zusammenspiel mit ihren Wirten zu analysieren. Übergeordnetes Ziel ist es, RNA-Phagen besser zu verstehen und herauszufinden, wie sie für die Entwicklung neuer antibakterieller Strategien genutzt werden können.
Unsere Forschung
Angesichts der zunehmenden Bedrohung durch antibiotikaresistente Bakterien sind neue Behandlungsmöglichkeiten für bakterielle Infektionen dringend erforderlich. Phagen als die natürlichen Gegenspieler von Bakterien könnten Abhilfe schaffen. Hierfür ist es jedoch entscheidend, geeignete Phagen zu identifizieren und ihre Funktionsweise zu entschlüsseln. RNA-Phagen sind – im Gegensatz zu DNA-Phagen – wenig erforscht. Ihre Charakterisierung wird dazu beitragen, das volle Potenzial von Phagen bei der Entwicklung neuartiger antimikrobieller Therapien auszuschöpfen und birgt die Chance, innovative molekulare Werkzeuge und Technologien zu konzipieren.
Die Arbeitsgruppe von Jens Hör untersucht die molekularen Grundlagen dieser RNA-Phagen im Kontext der Phagen-Wirt-Interaktionen. Ihre Forschung zielt darauf ab, zu ergründen, wie RNA-Phagen ihre Wirte während der Infektion unter ihre Kontrolle bringen und wie die Wirte sich gegen die eindringenden Phagen verteidigen. Außerdem isoliert das Hör-Labor neue RNA-Phagen und charakterisiert sie funktionell.
Die Gruppe nutzt sowohl klassische biochemische und molekularbiologische Methoden als auch modernste Hochdurchsatzverfahren, um die besondere Lebensweise von RNA-Phagen im Zusammenspiel mit ihren Wirten zu analysieren. Übergeordnetes Ziel ist es, RNA-Phagen besser zu verstehen und herauszufinden, wie sie für die Entwicklung neuer antibakterieller Strategien genutzt werden können.
Jun.-Prof. Dr. Jens Hör
RNA-Phagen stellen eine wenig erforschte Gruppe von Phagen dar. Unser Ziel ist es, ihre molekularen Grundlagen besser zu verstehen und herauszufinden, wie wir sie nutzen können, um neue antibakterielle Strategien zu entwickeln.

Jens Hör schloss 2013 sein Masterstudium in Lebens- und Gesundheitswissenschaften an der Universität Bonn ab und promovierte 2020 an der Universität Würzburg. Im Rahmen dieser Studien beschäftigte er sich mit der Analyse des globalen bakteriellen RNA-Protein-Komplexoms. Anschließend ging er als Postdoktorand an das Weizmann Institute of Science (Rehovot, Israel), wo er die Mechanismen der bakteriellen Anti-Phagen-Abwehr erforschte. Seit 2024 ist er Forschungsgruppenleiter am Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) und Juniorprofessur an der Universität Würzburg.
Ausgewählte Publikationen
Hör J, Wolf SG, Sorek R (2024). Bacteria conjugate ubiquitin-like proteins to interfere with phage assembly. Nature 631(8022):850-856. DOI: 10.1038/s41586-024-07616-5
Stokar-Avihail A, Fedorenko T, Hör J, Garb J, Leavitt A, Millman A, Shulman G, Wojtania N, Melamed S, Amitai G, Sorek R (2023). Discovery of phage determinants that confer sensitivity to bacterial immune systems. Cell 186(9):1863-1876.e16. DOI: 10.1016/j.cell.2023.02.029
Hör J, Jung J, Ðurica-Mitić S, Barquist L, Vogel J (2022). INRI-seq enables global cell-free analysis of translation initiation and off-target effects of antisense inhibitors. Nucleic Acids Research 50(22):e128. DOI: 10.1093/nar/gkac838
Hör J, Di Giorgio S, Gerovac M, Venturini E, Förstner KU, Vogel J (2020). Grad-seq shines light on unrecognized RNA and protein complexes in the model bacterium Escherichia coli. Nucleic Acids Research 48(16):9301-9319. DOI: 10.1093/nar/gkaa676
Hör J, Garriss G, Di Giorgio S, Hack LM, Vanselow JT, Förstner KU, Schlosser A, Henriques-Normark B, Vogel J (2020). Grad-seq in a Gram-positive bacterium reveals exonucleolytic sRNA activation in competence control. The EMBO Journal 39(9):e103852. DOI: 10.15252/embj.2019103852


