RNA-Biologie von bakteriellen Infektionen

Unsere Forschung
In unserer Arbeitsgruppe werden vier stark vernetzte Forschungsfelder bearbeitet:
Entdeckung funktioneller RNAs im Mikrobiom
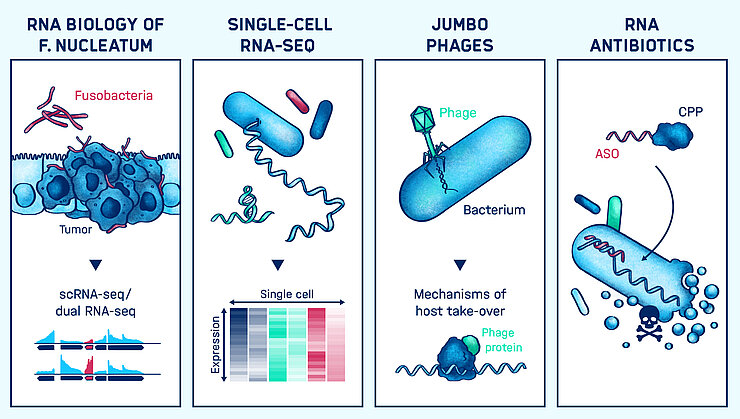
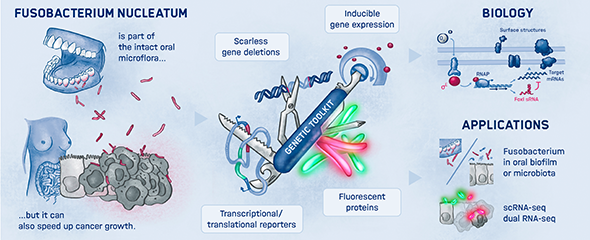
Die RNA-Biologie der meisten für die menschliche Gesundheit wichtigen Mikroorganismen ist noch weitgehend unverstanden, da die Hauptklassen nicht-kodierender RNA bislang nur in wenigen Bakterienarten untersucht wurden. Ein umfangreiches Verständnis der RNA-basierten Signalwege und Mechanismen ist jedoch von Nöten, damit das Potential von RNA als programmierbares Antibiotikum zur Behandlung bakterieller Infektionen ausgeschöpft werden kann. Daher haben wir in den letzten Jahren neue Methoden entwickelt, wie etwa differenzielles RNA-seq (Sharma et al. 2010 Nature) oder dual RNA-seq (Westermann et al. 2016 Nature), um zu verstehen, welche RNAs zu welchem Zeitpunkt im Verlauf einer Infektion exprimiert werden. Zudem können wir jetzt mit Grad-seq (Smirnov et al. 2016 PNAS) potentiell alle zellulären Transkripte anhand ihres biochemischen Verhaltens klassifizieren. Mit Hilfe dieser innovativen Verfahren beginnen wir nun, die Welt der RNA-Funktionen des menschlichen Mikrobioms zu verstehen.
Neue RNA-Bindungsproteine
Ihre funktionelle Aktivität erhalten nicht-kodierende RNA-Moleküle üblicherweise durch die Interaktion mit RNA-Bindeproteinen (RBP). In Eukaryoten sind RBP wichtige Bausteine der post-transkriptionellen Genregulierung; ihre Anzahl beläuft sich dort auf hunderte bis tausende verschiedene Proteine pro Organismus. Ironischerweise ist jedoch in Bakterien, die sich eigentlich am besten für molekulare Forschung eignen, bislang nur wenig über die Zahl an RBP und deren Funktionen bekannt. Obwohl die Genexpression in Bakterien zu großen Teilen auf der Ebene der RNA reguliert wird, beschränkt sich unser Wissen weitgehend auf das sRNA-Chaperon Hfq und den Translationshemmer CsrA. Nicht zuletzt hat jedoch die Entdeckung von CRISPR-Cas-Systemen deutlich gemacht, wie groß das Potential von RBP in in Mikroorganismen ist.
Kürzlich haben wir mit dem „osmoregulatorischen“ Protein ProQ ein neues, weit verbreitetes RBP entdeckt, welches hunderte zellulärer RNAs in Salmonella und E. coli bindet (Smirnov et al. 2016 PNAS). Unsere ersten Ergebnisse zeigen, dass ProQ an einem neuartigen Mechanismus zur Genregulierung am 3´-Ende von mRNAs beteiligt ist und die Struktur und nicht die Sequenz von RNA erkennt (Smirnov et al. 2017 EMBO J; Holmqvist et al. 2018 Molecular Cell). Außerdem untersuchen wir, wie die FinO-Domäne von ProQ-ähnlichen Proteinen RNA-Moleküle entweder hochspezifisch oder universell erkennt, wodurch wir grundlegende Prinzipien der RNA-Erkennung durch Proteine verstehen möchten.
Einzelzell-RNA-Biologie
Die Entwicklung der Einzelzell-RNA-Sequenzierung und die damit ermöglichte Analyse der Genexpressionsprofile individueller Zellen verändert derzeit die Sicht auf viele Aspekte der Biologie und hat bereits zur Entdeckung neuer Zelltypen und –funktionen geführt. Prinzipiell bietet diese Methode auch die Möglichkeit, die Variabilität zwischen einzelnen Bakterienzellen zu bestimmen. Aufgrund technischer Beschränkungen konnte bislang jedoch ausschließlich das Transkriptom eukaryotischer Zellen analysiert werden. Wir haben beispielsweise untersucht, wie sich heterogenes intrazelluläres Wachstum von Salmonella auf die Polarisation infizierter Makrophagen auswirkt (Saliba et al. 2017 Nature Microbiology).
Als große Herausforderung sehen wir die Einzelzell-RNA-Sequenzierung in Bakterien an. Faktoren dafür weshalb das bisher kaum möglich ist sind die viel geringere RNA-Menge einzelner Bakterien und der fehlende polyA-Schwanz im Gegensatz zu eukaryontischen Transkripten. Transkriptomdaten einzelner Bakterienzellen würden Einblicke in die Bildung von Resistenzen gegenüber Antibiotika und die Bildung von Persistenzformen gewähren. Außerdem wäre dies ein wichtiger Baustein für die duale Einzelzell-RNA-Sequenzierung, um simultan Interaktionen zwischen individuellen Wirts- und Pathogenzellen zu untersuchen. Unser langfristiges Ziel ist die Entwicklung von Hochdurchsatz-Methoden für RNA-Biochemie einzelner Bakterienzellen, um die Mechanismen der post-transkriptionellen Genregulation untersuchen zu können.
RNA als Instrument zum Manipulieren des Mikobioms
Unser Körper beherbergt eine Vielzahl an Bakterien, welche zusammen das menschliche Mikrobiom bilden. Allein unser Darm ist von mehr als tausend verschiedenen Bakterienarten besiedelt. Damit wir deren individuelle oder synergistische Beiträge zu unserer Gesundheit verstehen, muss es uns gelingen, die Funktion einzelner Bakterienarten gezielt zu beeinflussen. Derzeit verfügbare Antibiotika bieten dieses Maß an Selektivität jedoch nicht.
Unser Ziel ist eine präzise Manipulation des Mikrobiom durch “Programmierbare Antibiotika” in der Form von RNA-gerichteten Antisense-Oligonukleotiden (ASOs). Dieselbe Methode böte sich auch als Alternative zu herkömmlichen Antibiotika bei der Bekämpfung von antibiotikaresistenten Krankheitserregern an. Die ASOs sind dabei an kleine Peptide gekoppelt, mit deren Hilfe sie in Bakterienzellen eindringen und die Transkripte essentieller Gene ausschalten. Einige Fragen dieser vielversprechenden Methode müssen jedoch noch geklärt werden: Wie wird gewährleistet, dass ASOs ausschließlich artspezifisch funktionieren? Wie können mögliche Nebeneffekte minimiert werden? Welche Peptide eignen sich besonders als Vehikel in die Zellen? Welche Reaktionen können in der Wirtszelle auftreten und welche Resistenz-Mechanismen bilden sich in den Bakterien? Da nicht mit einer Universallösung für sämtliche Szenarien zu rechnen ist, kann das Projekt nur durch die starke Vernetzung von Expertenwissen aus den Bereichen RNA-Biologie, Mikrobiologie, Immunologie, Einzelzell-Biologie und Pharmazie erfolgreich sein.
Unsere Forschung
In unserer Arbeitsgruppe werden vier stark vernetzte Forschungsfelder bearbeitet:
Entdeckung funktioneller RNAs im Mikrobiom
Die RNA-Biologie der meisten für die menschliche Gesundheit wichtigen Mikroorganismen ist noch weitgehend unverstanden, da die Hauptklassen nicht-kodierender RNA bislang nur in wenigen Bakterienarten untersucht wurden. Ein umfangreiches Verständnis der RNA-basierten Signalwege und Mechanismen ist jedoch von Nöten, damit das Potential von RNA als programmierbares Antibiotikum zur Behandlung bakterieller Infektionen ausgeschöpft werden kann. Daher haben wir in den letzten Jahren neue Methoden entwickelt, wie etwa differenzielles RNA-seq (Sharma et al. 2010 Nature) oder dual RNA-seq (Westermann et al. 2016 Nature), um zu verstehen, welche RNAs zu welchem Zeitpunkt im Verlauf einer Infektion exprimiert werden. Zudem können wir jetzt mit Grad-seq (Smirnov et al. 2016 PNAS) potentiell alle zellulären Transkripte anhand ihres biochemischen Verhaltens klassifizieren. Mit Hilfe dieser innovativen Verfahren beginnen wir nun, die Welt der RNA-Funktionen des menschlichen Mikrobioms zu verstehen.
Neue RNA-Bindungsproteine
Ihre funktionelle Aktivität erhalten nicht-kodierende RNA-Moleküle üblicherweise durch die Interaktion mit RNA-Bindeproteinen (RBP). In Eukaryoten sind RBP wichtige Bausteine der post-transkriptionellen Genregulierung; ihre Anzahl beläuft sich dort auf hunderte bis tausende verschiedene Proteine pro Organismus. Ironischerweise ist jedoch in Bakterien, die sich eigentlich am besten für molekulare Forschung eignen, bislang nur wenig über die Zahl an RBP und deren Funktionen bekannt. Obwohl die Genexpression in Bakterien zu großen Teilen auf der Ebene der RNA reguliert wird, beschränkt sich unser Wissen weitgehend auf das sRNA-Chaperon Hfq und den Translationshemmer CsrA. Nicht zuletzt hat jedoch die Entdeckung von CRISPR-Cas-Systemen deutlich gemacht, wie groß das Potential von RBP in in Mikroorganismen ist.
Kürzlich haben wir mit dem „osmoregulatorischen“ Protein ProQ ein neues, weit verbreitetes RBP entdeckt, welches hunderte zellulärer RNAs in Salmonella und E. coli bindet (Smirnov et al. 2016 PNAS). Unsere ersten Ergebnisse zeigen, dass ProQ an einem neuartigen Mechanismus zur Genregulierung am 3´-Ende von mRNAs beteiligt ist und die Struktur und nicht die Sequenz von RNA erkennt (Smirnov et al. 2017 EMBO J; Holmqvist et al. 2018 Molecular Cell). Außerdem untersuchen wir, wie die FinO-Domäne von ProQ-ähnlichen Proteinen RNA-Moleküle entweder hochspezifisch oder universell erkennt, wodurch wir grundlegende Prinzipien der RNA-Erkennung durch Proteine verstehen möchten.
Einzelzell-RNA-Biologie
Die Entwicklung der Einzelzell-RNA-Sequenzierung und die damit ermöglichte Analyse der Genexpressionsprofile individueller Zellen verändert derzeit die Sicht auf viele Aspekte der Biologie und hat bereits zur Entdeckung neuer Zelltypen und –funktionen geführt. Prinzipiell bietet diese Methode auch die Möglichkeit, die Variabilität zwischen einzelnen Bakterienzellen zu bestimmen. Aufgrund technischer Beschränkungen konnte bislang jedoch ausschließlich das Transkriptom eukaryotischer Zellen analysiert werden. Wir haben beispielsweise untersucht, wie sich heterogenes intrazelluläres Wachstum von Salmonella auf die Polarisation infizierter Makrophagen auswirkt (Saliba et al. 2017 Nature Microbiology).
Als große Herausforderung sehen wir die Einzelzell-RNA-Sequenzierung in Bakterien an. Faktoren dafür weshalb das bisher kaum möglich ist sind die viel geringere RNA-Menge einzelner Bakterien und der fehlende polyA-Schwanz im Gegensatz zu eukaryontischen Transkripten. Transkriptomdaten einzelner Bakterienzellen würden Einblicke in die Bildung von Resistenzen gegenüber Antibiotika und die Bildung von Persistenzformen gewähren. Außerdem wäre dies ein wichtiger Baustein für die duale Einzelzell-RNA-Sequenzierung, um simultan Interaktionen zwischen individuellen Wirts- und Pathogenzellen zu untersuchen. Unser langfristiges Ziel ist die Entwicklung von Hochdurchsatz-Methoden für RNA-Biochemie einzelner Bakterienzellen, um die Mechanismen der post-transkriptionellen Genregulation untersuchen zu können.
RNA als Instrument zum Manipulieren des Mikobioms
Unser Körper beherbergt eine Vielzahl an Bakterien, welche zusammen das menschliche Mikrobiom bilden. Allein unser Darm ist von mehr als tausend verschiedenen Bakterienarten besiedelt. Damit wir deren individuelle oder synergistische Beiträge zu unserer Gesundheit verstehen, muss es uns gelingen, die Funktion einzelner Bakterienarten gezielt zu beeinflussen. Derzeit verfügbare Antibiotika bieten dieses Maß an Selektivität jedoch nicht.
Unser Ziel ist eine präzise Manipulation des Mikrobiom durch “Programmierbare Antibiotika” in der Form von RNA-gerichteten Antisense-Oligonukleotiden (ASOs). Dieselbe Methode böte sich auch als Alternative zu herkömmlichen Antibiotika bei der Bekämpfung von antibiotikaresistenten Krankheitserregern an. Die ASOs sind dabei an kleine Peptide gekoppelt, mit deren Hilfe sie in Bakterienzellen eindringen und die Transkripte essentieller Gene ausschalten. Einige Fragen dieser vielversprechenden Methode müssen jedoch noch geklärt werden: Wie wird gewährleistet, dass ASOs ausschließlich artspezifisch funktionieren? Wie können mögliche Nebeneffekte minimiert werden? Welche Peptide eignen sich besonders als Vehikel in die Zellen? Welche Reaktionen können in der Wirtszelle auftreten und welche Resistenz-Mechanismen bilden sich in den Bakterien? Da nicht mit einer Universallösung für sämtliche Szenarien zu rechnen ist, kann das Projekt nur durch die starke Vernetzung von Expertenwissen aus den Bereichen RNA-Biologie, Mikrobiologie, Immunologie, Einzelzell-Biologie und Pharmazie erfolgreich sein.
Prof. Jörg Vogel
Die Bedeutung von RNA-Molekülen bei Infektionsprozessen ist bis vor kurzem unterschätzt worden. Dagegen wissen wir heute, dass RNA mit einer Vielzahl von Molekülen der Wirtszelle und des Krankheitserregers interagiert. Um diese Wechselwirkungen medizinisch nutzbar zu machen, verfolgen wir am HIRI ein integratives Konzept unter der Verwendung von hochmodernen Technologien zur RNA-Analyse.

Professor Jörg Vogel studierte Biochemie an der Berliner Humboldt-Universität und am Imperial College (London, UK). 1999 promovierte er an der Humboldt-Universität über „Group II intron splicing“. Als Postdoc machte er von 2000 bis 2001 Station an der Universität Upsala (Schweden) und von 2002 bis 2003 als EMBO-Stipendiat an der Hebräischen Universität in Jerusalem (Israel). Von 2004 bis 2010 war er unabhängiger Nachwuchsgruppenleiter am Max-Planck-Institut für Molekulare Infektionsbiologie, seit 2009 ist er Lehrstuhlinhaber und Direktor des Instituts für Molekulare Infektionsbiologie an der Universität Würzburg. Seit 2017 ist er ebenfalls Gründungsdirektor des Würzburger Helmholtz-Instituts für RNA-basierte Infektionsforschung.
Jörg Vogel ist ein gewähltes Mitglied von EMBO, der Deutschen Nationalen Akademie der Wissenschaften und der Europäischen sowie der Amerikanischen Akademie der Mikrobiologie. Für seine integrativen Arbeiten in der RNA-Biologie und Infektionsforschung erhielt er 2017 den renommierten Leibniz-Preis.
Ausgewählte Publikationen
Sharma CM, Hoffmann S, Darfeuille F, Reignier J, Findeiß S, Sittka A, Chabas S, Reiche K, Hackermüller J, Reinhardt R, Stadler PF, Vogel J (2010) The primary transcriptome of the major human pathogen Helicobacter pylori. Nature 464 250-255
Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao Y, Pirzada ZA, Eckert MR, Vogel J, Charpentier E (2011) CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature 471: 602-7
Papenfort K, Sun Y, Miyakoshi M, Vanderpool CK, Vogel J (2013) Small RNA-mediated activation of sugar phosphatase mRNA regulates glucose homeostasis. Cell 153: 426–437
Smirnov A, Förstner KU, Holmqvist E, Otto A, Günster R, Becher D, Reinhardt R, Vogel J (2016) Grad-seq guides the discovery of ProQ as a major small RNA binding protein. PNAS 113: 11591-6
Westermann AJ, Förstner KU, Amman F, Barquist L, Chao Y, Schulte LN, Müller L, Reinhardt R, Stadler PF, Vogel J (2016) Dual RNA-seq unveils noncoding RNA functions in host-pathogen interactions. Nature 529: 496-501
Technologieangebote
Folgende Technologien wurden von der Abteilung "RNA-Biologie bakterieller Infektionen" entwickelt und zum Patent angemeldet:
Novel ASO-Based mRNA Silencing Technology for Phage-Host Interaction Research