Strukturelle Infektionsbiologie

Unsere Forschung
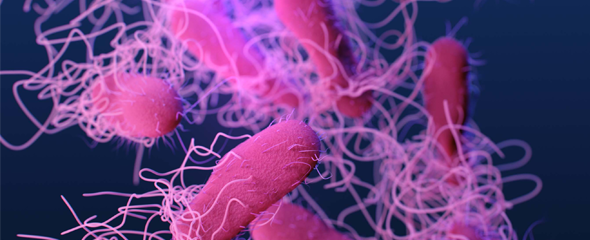
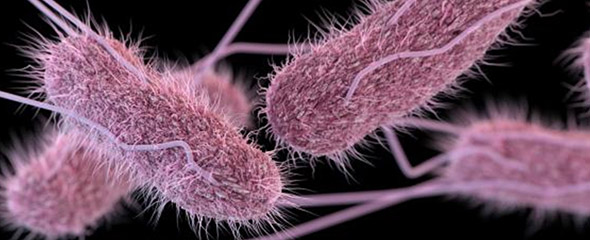
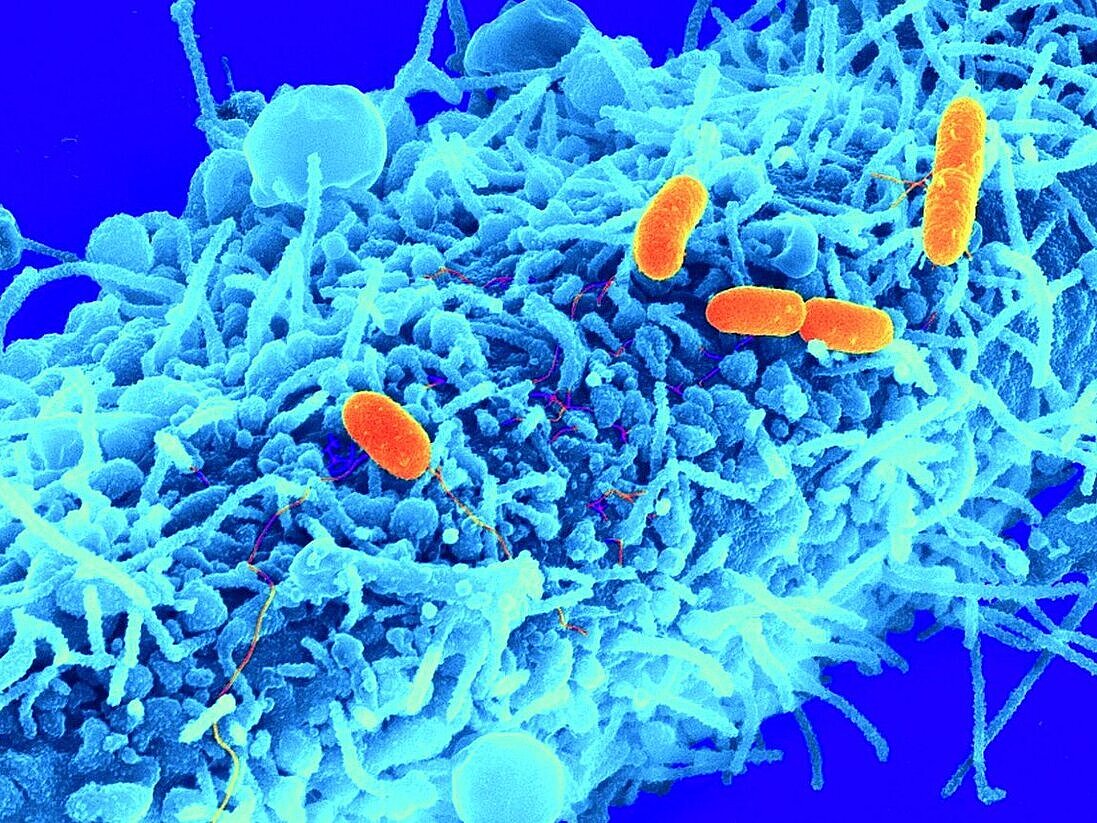
Krankheitserreger manipulieren humane Wirtszellen für einen möglichst effizienten Infektionsverlauf. Zu diesem Zweck setzen verschiedenste bakterielle Erreger gezielt Virulenzfaktoren frei, die u. a. deren Invasion in Wirtszellen erleichtert oder die Immunantwort des Wirtes unterdrückt.
Infektionsstrategien identifizieren
Mittels genomischer und proteomischer Analysen konnten in den letzten Jahren viele dieser Faktoren identifiziert und die darauf basierenden Infektionsstrategien entschlüsselt werden. In vielen gramnegativen Bakterien ist ein großer, membranständiger Proteinkomplex, dass sogenannte Typ-3-Sekretionssystem (T3SS) essentiell für die kontrollierte Freisetzung verschiedenster Virulenzfaktoren. Dieser aus vielen Untereinheiten bestehende makromolekulare Komplex ist wie eine Spritze geformt und zieht sich durch die gesamte bakterielle Doppelmembran. Die Virulenzproteine werden aktiv und nach einem bestimmten zeitlichen und räumlichen Muster durch das T3SS transportiert.
Unser primäres Ziel ist es, die Struktur und Funktion des T3SS zu entschlüsseln sowie die Transportdynamik und die damit verbundenen Regulationsmechanismen aufzuklären.
Strukturbiologische Techniken und biophysikalische Methoden
Wir nutzen verschiedenste molekularbiologische und proteinbiochemische Techniken sowie zelluläre Untersuchungsmethoden für unsere Forschung. Darüber hinaus verwenden wir modernste strukturbiologische Techniken (Röntgenstrukturanalyse, Elektronen- und Fluoreszenzmikroskopie) in Kombination mit verschiedenen biophysikalischen Methoden (Lichtstreuung, Kalorimetrie, CD- und Fluoreszenzspektroskopie) für die möglichst umfassende Struktur-/Funktionsanalyse des T3SS im Verlaufe bakterieller Infektionen.
Unser übergeordnetes Ziel bei der Entschlüsselung der molekularen Mechanismen der Wirtszellinvasion liegt darin, einen Beitrag bei der Entwicklung rationaler Therapieansätze sowie von effizienten Antiinfektiva gegen gramnegative Bakterien zu leisten. Die Weltgesundheitsorganisation schätzt, dass mehr als 100 Millionen Menschen weltweit jedes Jahr von Krankheiten, ausgelöst von gramnegativen Bakterien wie Gastroenteritis (Shigella flexneri), Typhus-Fieber (Salmonella typhi), Lebensmittelvergiftungen (Escherichia coli spp) und Beulenpest (Yersinia pestis), betroffen sind und diese sogar einige Millionen Tote fordert.
Unsere Forschung
Krankheitserreger manipulieren humane Wirtszellen für einen möglichst effizienten Infektionsverlauf. Zu diesem Zweck setzen verschiedenste bakterielle Erreger gezielt Virulenzfaktoren frei, die u. a. deren Invasion in Wirtszellen erleichtert oder die Immunantwort des Wirtes unterdrückt.
Infektionsstrategien identifizieren
Mittels genomischer und proteomischer Analysen konnten in den letzten Jahren viele dieser Faktoren identifiziert und die darauf basierenden Infektionsstrategien entschlüsselt werden. In vielen gramnegativen Bakterien ist ein großer, membranständiger Proteinkomplex, dass sogenannte Typ-3-Sekretionssystem (T3SS) essentiell für die kontrollierte Freisetzung verschiedenster Virulenzfaktoren. Dieser aus vielen Untereinheiten bestehende makromolekulare Komplex ist wie eine Spritze geformt und zieht sich durch die gesamte bakterielle Doppelmembran. Die Virulenzproteine werden aktiv und nach einem bestimmten zeitlichen und räumlichen Muster durch das T3SS transportiert.
Unser primäres Ziel ist es, die Struktur und Funktion des T3SS zu entschlüsseln sowie die Transportdynamik und die damit verbundenen Regulationsmechanismen aufzuklären.
Strukturbiologische Techniken und biophysikalische Methoden
Wir nutzen verschiedenste molekularbiologische und proteinbiochemische Techniken sowie zelluläre Untersuchungsmethoden für unsere Forschung. Darüber hinaus verwenden wir modernste strukturbiologische Techniken (Röntgenstrukturanalyse, Elektronen- und Fluoreszenzmikroskopie) in Kombination mit verschiedenen biophysikalischen Methoden (Lichtstreuung, Kalorimetrie, CD- und Fluoreszenzspektroskopie) für die möglichst umfassende Struktur-/Funktionsanalyse des T3SS im Verlaufe bakterieller Infektionen.
Unser übergeordnetes Ziel bei der Entschlüsselung der molekularen Mechanismen der Wirtszellinvasion liegt darin, einen Beitrag bei der Entwicklung rationaler Therapieansätze sowie von effizienten Antiinfektiva gegen gramnegative Bakterien zu leisten. Die Weltgesundheitsorganisation schätzt, dass mehr als 100 Millionen Menschen weltweit jedes Jahr von Krankheiten, ausgelöst von gramnegativen Bakterien wie Gastroenteritis (Shigella flexneri), Typhus-Fieber (Salmonella typhi), Lebensmittelvergiftungen (Escherichia coli spp) und Beulenpest (Yersinia pestis), betroffen sind und diese sogar einige Millionen Tote fordert.
Prof. Dr. Michael Kolbe
Um die molekularen Virulenzmechanismen in der Pathogenese bakterieller Infektionen zu verstehen, bedarf es der Integration komplementärer Technologien der molekularen Genetik, der Mikrobiologie sowie biophysikalischer Methoden. Das CSSB auf dem Campus des DESY bietet exzellente Bedingungen für einen interdisziplinären Forschungsansatz, da es Infektionsbiologen mit modernsten Instrumenten ausstattet und fortlaufend neue spektroskopische Techniken entwickelt.

Michael Kolbe studierte Chemie an den Universitäten Paderborn und Hamburg. Er promovierte anschließend am Max-Planck-Institut für Biochemie und der Ludwig-Maximilians-Universität in München bei Dieter Oesterhelt mit einer Arbeit zur Struktur und Funktion der Chloridpumpe Halorhodopsin. Im Anschluss arbeitete Michael Kolbe als Post-Doc am Max-Delbrück Centrum in Berlin unter anderem an der Struktur von Ionenkanälen. Hier begann er sich erstmals für Proteintransporter zu interessieren, die eine wichtige Rolle bei bakteriellen Infektionen von Wirtszellen spielen.
Im Anschluss wechselte er als Nachwuchsgruppenleiter an das Max-Planck-Institut für Infektionsbiologie, wo er seine Arbeiten an Typ 3 Sekretionssystemen vertiefte. Die Forschung seiner Gruppe für Strukturelle Systembiologie wird seit 2013 durch das ERC gefördert.
Mit Beginn des Jahres 2015 ist Michael Kolbe Professor an der Universität Hamburg und Leiter der Abteilung für Strukturelle Infektionsbiologie des HZI. Die Abteilung wird Anfang 2017 ihre Räume im neu gegründeten Center for Structural Systems Biology auf dem DESY Campus in Hamburg beziehen.
Ausgewählte Publikationen
Human aryl hydrogen receptor detects virulence factors from bacterial pathogens
Moura-Alves P et al, Aryl hydrogen receptor senses bacterial pigmented virulence factors and orchestrates antibacterial defenses, Nature, 512: 387-392 (2014)
Shigella flexneri type 3 secretion system caught in the act
Dohlich K, Brotcke Zumsteg A, Goosmann C, Kolbe M, A Substrate-Fusion Protein is Trapped inside the Type III Secretion System Channel in Shigella flexneri, PLoS Pathogens, e003881 (2014)
3-dimensional model of the type 3 secretion needle in Salmonella typhimurium
Loquet A Sgourakis NG, Gupta R, Giller K, Riedel D, Goosmann C, Griesinger C, Kolbe* M, Baker D, Becker S, Lange* A, Atomic model of the type III secretion system needle, Nature, 486: 276-279 (2012)
Structure and mechanism of type 3 secretion system needle assembly
Poyraz O, Schmidt H, Seidel K, Delissen F, Ader C, Tenenboim H, Goosmann C, Laube B, Thünemann AF, Zychlinsky A, Baldus M, Lange A, Griesinger C, Kolbe M, Protein refolding is required for assembly of the type three secretion needle. Nat. Struct. Mol. Biol., 17: 788-792 (2010)
Structural based prediction of a conserved effector-chaperone motif for type 3 secretion
Lunelli, M, Lokareddy, RK, Zychlinsky, A, Kolbe, M, IpaB-IpgC interaction defines binding motif for type III secretion translocator. Proc. Natl. Acad. Sci., 106: 9661-9666 (2009)
Publikationen
Newsroom
Sie haben Interesse an einer Bachelor- oder Masterarbeit? Wir freuen uns auf Ihre Anfrage!