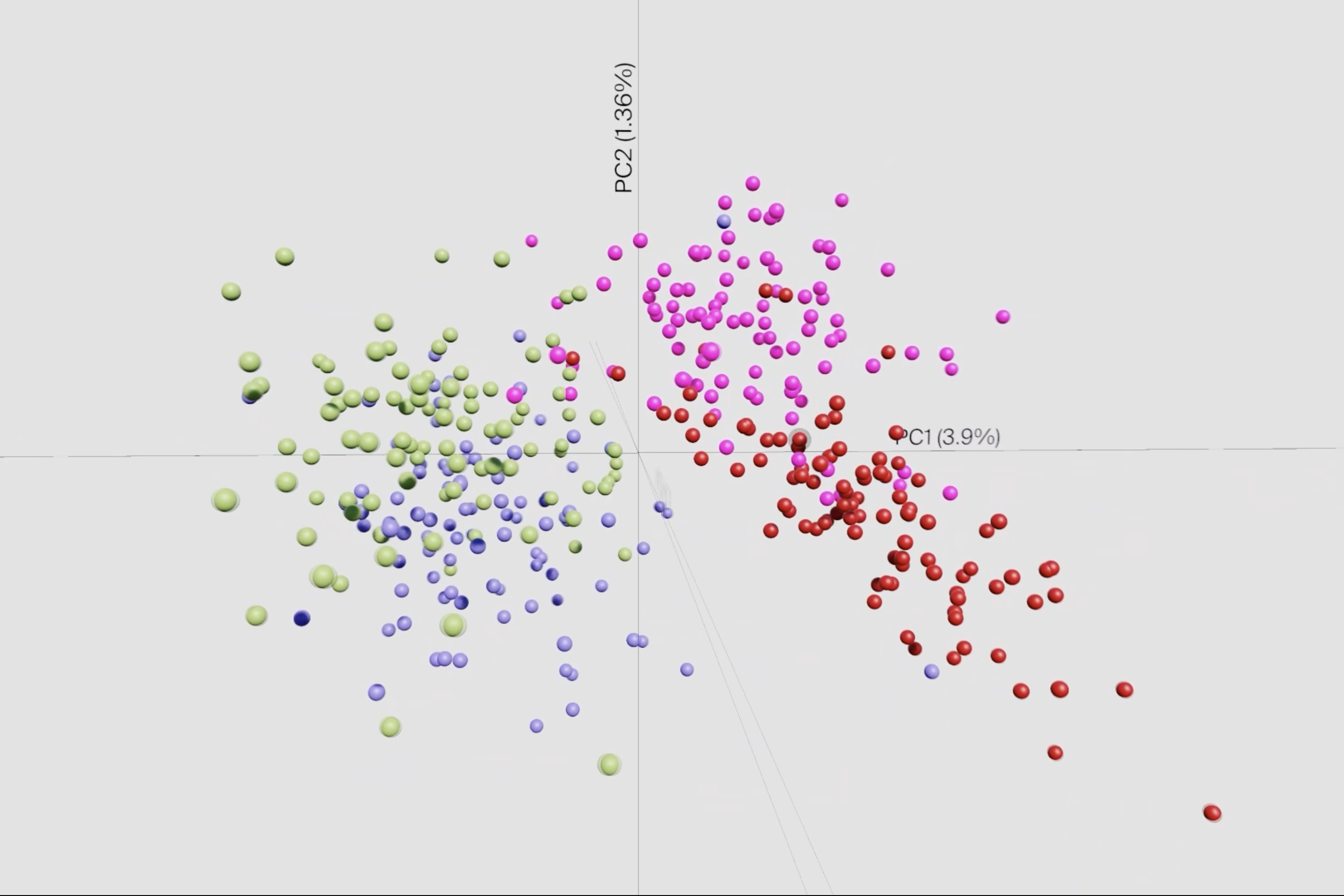
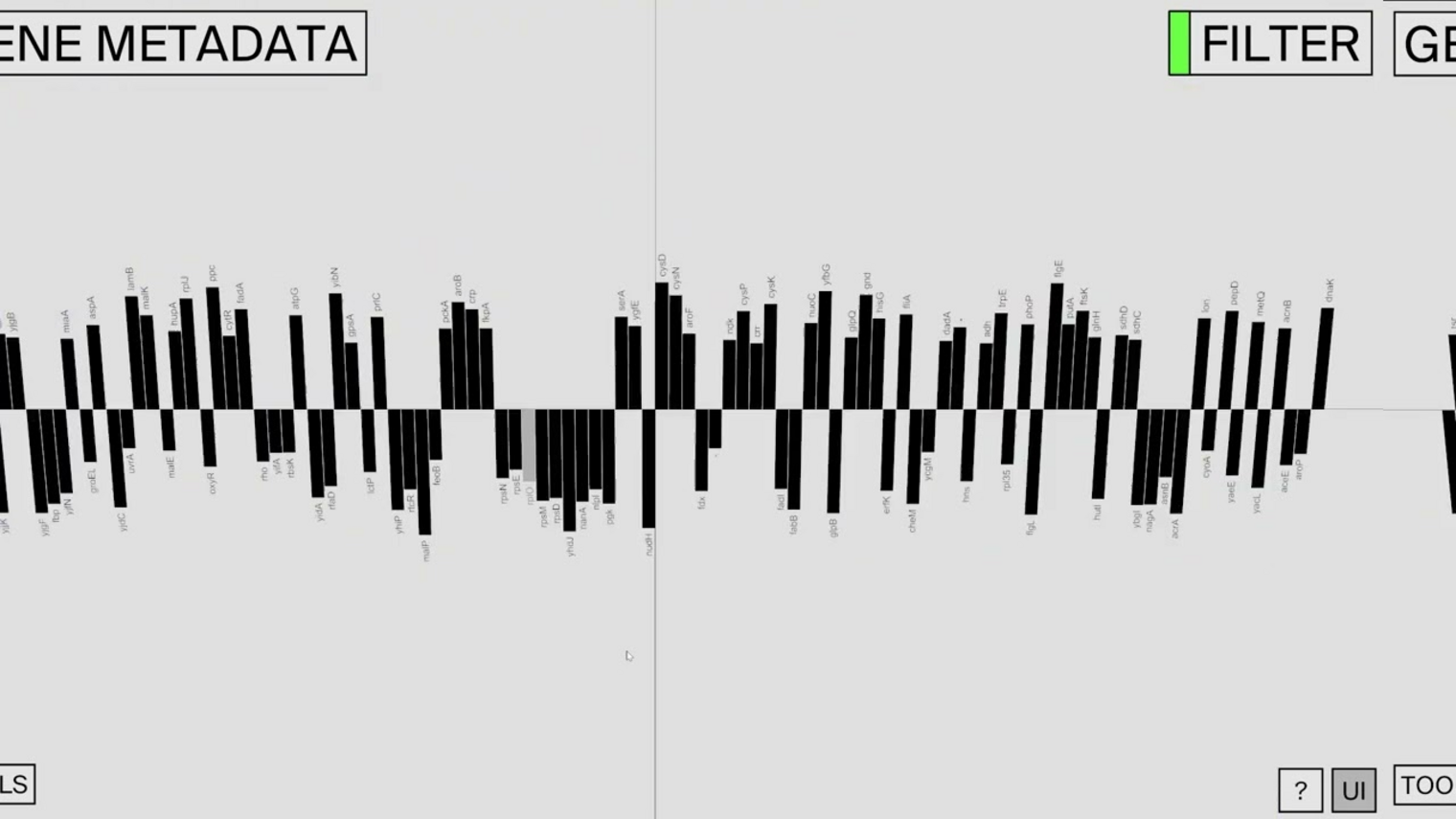
Antibiotikaresistenzen sind weltweit auf dem Vormarsch und stellen das Gesundheitswesen vor große Herausforderungen. Es gilt also herauszufinden, wie und warum manche Bakterien Resistenzen entwickeln. Dafür ist es wichtig, die zugrundeliegenden Prozesse zu verstehen. Unterschiede in der bakteriellen Genexpression, bei der die Information eines Gens abgelesen wird, könnten Hinweise liefern. Als bedeutendes Hilfsmittel für deren Analyse hat sich die bakterielle Einzelzell-RNA-Sequenzierung, kurz „scRNA-seq“ (von engl. single-cell RNA sequencing), erwiesen. Diese Technologie liefert ein genaues Abbild der Expression von Genen in einer einzelnen Zelle zu einem bestimmten Zeitpunkt. Sie ermöglicht so Rückschlüsse auf die molekularen Grundlagen der Zellaktivität – mit einem großen Manko: Sie erzeugt riesige Datensätze, die für Forschende schwer zu visualisieren und zu analysieren sind. Entsprechend groß ist der Bedarf an neuen Ansätzen und Werkzeugen, um diese Informationen zu veranschaulichen und verstehen.
Eine solche Anwendung haben nun Forschende vom Würzburger Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI), einem Standort des Braunschweiger Helmholtz-Zentrums für Infektionsforschung (HZI) in Kooperation mit der Julius-Maximilians-Universität Würzburg (JMU), und Designer der Technischen Hochschule Würzburg-Schweinfurt (THWS) gemeinsam entwickelt. Deren Desktop-Anwendung, genannt sCIRCLE (single-Cell Interactive Real-time Computer visualization for Low-dimensional Exploration), ermöglicht eine interaktive 3D-Visualisierung von Daten, die mithilfe von scRNA-seq erzeugt wurden. Damit können Nutzer:innen Daten einzelner Zellen, inklusive Metadaten, aus verschiedenen Blickwinkeln und in Echtzeit betrachten.