Der menschliche Darm beherbergt rund 100 Billionen Bakterien, also etwa zehnmal so viel wie der gesamte Körper Zellen hat. Diese innere Menagerie, das Mikrobiom, wiegt circa ein Kilogramm und besteht aus mehr als 500 verschiedenen Arten. Zudem ist sie individuell zusammengesetzt – jeder Mensch hat seinen ganz eigenen Zoo.
Das Mikrobiom hilft dem Wirt auf vielfältige Weise, etwa beim Verdauen bestimmter Bestandteile der Nahrung, zum Beispiel Ballaststoffe, die der Wirt selbst nicht zersetzen kann. Außerdem stellen die Bakterien des Mikrobioms viele B-Vitamine her – zwar für ihren eigenen Bedarf, aber auch der Wirt nimmt diese Vitamine auf.
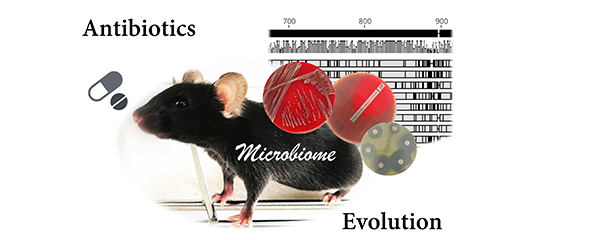
Das Mikrobiom wechselwirkt zudem mit dem Immunsystem und instruiert Immunantworten im Darm, aber auch im übrigen Körper bis zum Gehirn. Ein intaktes Mikrobiom ist daher wichtig für die Gesundheit, doch es reagiert sehr sensibel auf äußere Einflüsse wie bestimmte Nahrungsbestandteile und Medikamente. Insbesondere Antibiotika, die essenziell für die Bekämpfung von lebensbedrohlichen bakteriellen Infektionen sind, wirken auch auf die „guten“ Bakterien und können bis zu einem Drittel der Darmbewohner töten. Diese Schädigung ist normalerweise kurzlebig, das Mikrobiom erholt sich in zwei bis vier Wochen. Bei kranken oder älteren Menschen und Kindern kann die Erholungsphase jedoch länger dauern. Neueste Studien deuten darauf hin, dass eine vermehrte Antibiotikagabe vor allem bei Kindern unter drei Jahren zu einer langfristigen Beeinträchtigung des Mikrobioms führen kann – eine Zwickmühle, denn gerade in dieser Lebensphase greifen Ärzte häufig zu Antibiotika, etwa bei einer Mittelohrentzündung. Allerdings gibt es in dem Fall auch keine Alternative.
Besonders in Krankenhäusern geht der hohe Antibiotikaeinsatz mit einem erhöhten Risiko für den Erwerb von Krankheitserregern einher. Ist das Mikrobiom geschädigt, nutzen das Erreger wie Clostridium difficile, Staphylococcus aureus, Klebsiella pneumoniae oder enteropathogene Escherichia coli aus, um sich im Darm anzusiedeln. Vor allem C. difficile ist inzwischen ein großes Problem in Krankenhäusern, da es umweltresistente Sporen bildet, die für Monate außerhalb des Körpers überdauern können. Nach der Aufnahme durch Antibiotika-behandelte Patienten keimen diese Sporen aus und produzieren Giftstoffe, die die Darmwand lähmen und im schlimmsten Fall zu einer ballonartigen, mitunter lebensbedrohlichen Ausweitung des Dickdarms führen.
Diese teils heftigen Nebenwirkungen verdeutlichen, dass Antibiotika keine Spielzeuge sind und – zumindest für einige ihrer Einsatzgebiete – Alternativen wünschenswert wären. Ein möglicher Ansatzpunkt ist das Mikrobiom selbst: Studien in den USA und den Niederlanden zeigen vermehrt, dass zur Behandlung von bestimmten gastrointestinalen bakteriellen Infektionen Stuhltransplantationen einen ähnlichen Effekt haben können wie Antibiotika: Die Besiedlung mit „guten“ Darmbakterien führt zu einer Verdrängung der Erreger und kann Neuinfektionen verhindern. Allerdings ist das noch ein Zukunftsmodell, denn der Transfer einer Vielzahl von bekannten und unbekannten Bakterien birgt mögliche langfristige Risiken für den Empfänger. Deshalb erforschen Wissenschaftler derzeit die Gabe definierter Bakterienarten oder komplett andere Behandlungsansätze wie die Gabe von Erreger-spezifischen Phagen, die das Mikrobiom nicht angreifen.
Entscheidend für neue Therapieansätze ist es, die Wechselwirkungen zwischen Mikrobiom, Immunsystem und Pathogenen zu verstehen. Dabei ist es gar nicht so einfach, das Mikrobiom genau zu untersuchen: Eine Sequenzierung beispielsweise liefert eine riesige Datenmenge aller Bakterien, bezeichnet als Metagenom, aus der die Informationen über die einzelnen Arten herausgefiltert werden müssen. Dafür gibt es zwar viele Methoden, welche sich aber für die jeweilige Fragestellung am besten eignet, ist oft unklar.
Um diese Probleme zu lösen, hat ein internationales Wissenschaftlerteam – darunter auch Alice McHardy mit ihrer HZI-Abteilung „Bioinformatik der Infektionsforschung“ am BRICS – die Initiative „Critical Assessment of Metagenome Interpretation (CAMI)“ gegründet. In einem Wettbewerb können Wissenschaftler ihre bioinformatischen Methoden an verschiedenen Datensätzen testen und sie so evaluieren. „Das Ziel von CAMI ist es, die Analysemethoden für Metagenomdaten einheitlich und mit biologisch relevanten Massen zu prüfen und Standards zu entwickeln, welche Methode bei welcher wissenschaftlichen Fragestellung eingesetzt werden sollte“, sagt Alice McHardy.